Équipe Ait-Si-Ali
Dynamique épigénétique et différenciation cellulaire

Collaborateurs de l’équipe au 14 février 2025
© Slimane Aït-Si-Ali, 2025
Synopsis
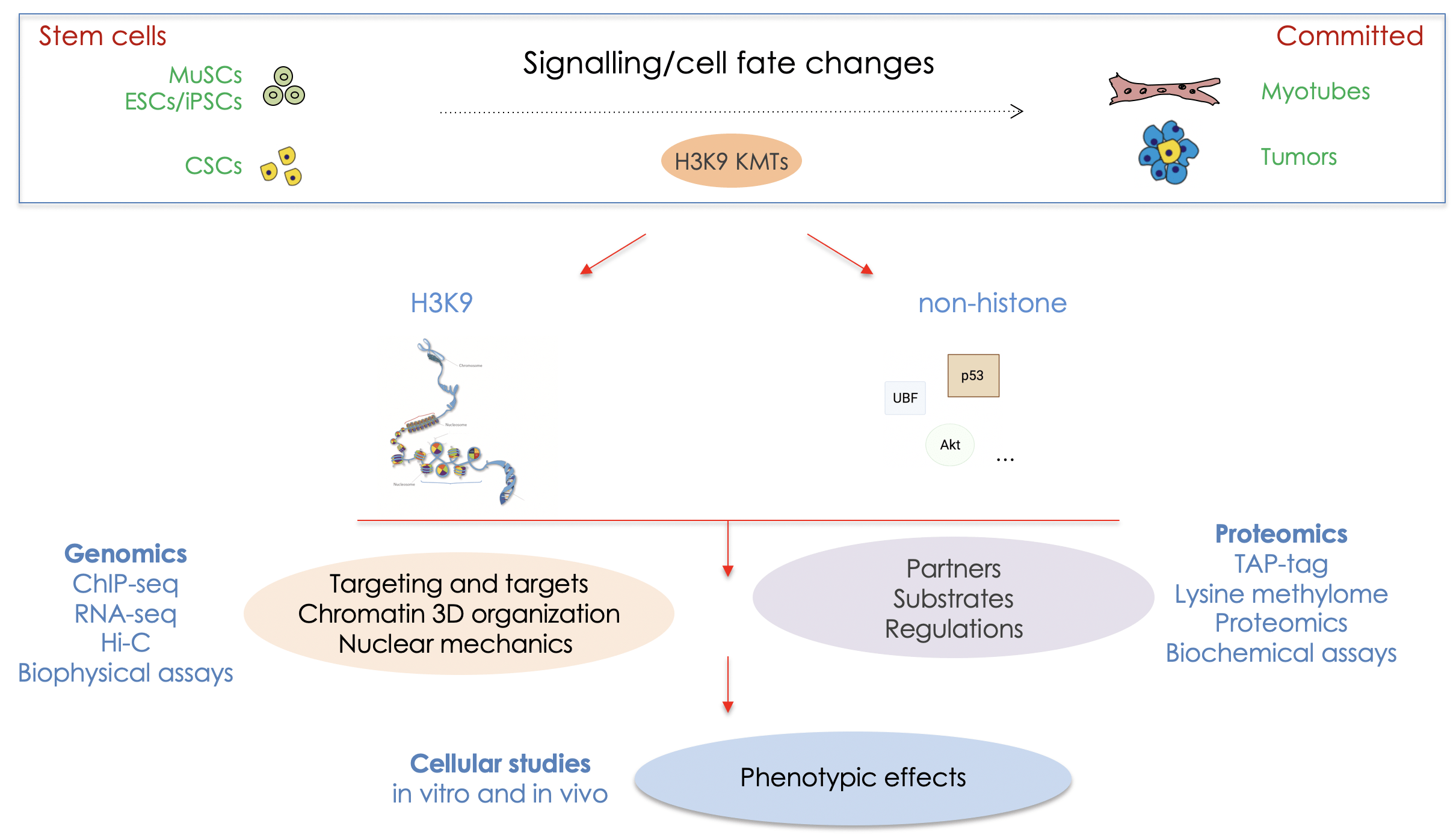
Un des mécanismes épigénétiques centraux est la méthylation/déméthylation de lysine des histones. La méthylation d’histones sur résidus lysines influe sur la structure de la chromatine et elle est au centre des mécanismes régulant de manière stable et héritable l’expression des gènes. La méthylation des histones peut être aussi bien associée à des domaines actifs que des domaines inactifs de la chromatine, selon le résidu lysine ciblé. La méthylation de l’histone H3 sur la lysine 9 (H3K9), tout comme sur la lysine 27 (H3K27), est caractéristique d’une forme de chromatine répressive. Les enzymes régulant la méthylation des lysines au niveau des histones sont les lysines méthyltransférases (KMTs) et lysines déméthylases (KDMs), la plupart ont la capacité de modifier également des substrats non histones. Elles sont hautement spécifiques des résidus cibles au niveau des histones et du degré de méthylation des lysines, mono, di ou triméthylés. Environ 50 KMTs et 30 KDMs ont été décrites chez les mammifères. Nous nous intéressons particulièrement aux KMTs spécifiques de H3K9.
OBJECTIFS PRINCIPAUX : Contribution de la méthylation de lysines par les KMTs de H3K9 dans l’établissement et la maintenance de l’identité cellulaire
Décrypter les mécanismes moléculaires dans les cellules souches lors des modifications du lignage cellulaire, plus spécifiquement la différenciation terminale, est un des objectifs fondamentaux de la médecine moderne. La capacité de modifier un état cellulaire est une approche prometteuse pour la médecine régénérative.
Notre groupe cherche à élucider les mécanismes moléculaires gouvernant la régulation de l’expression génique normale pendant des changements de destin cellulaire. En particulier, nous nous intéressons au rôle de la méthylation des lysines et plus spécifiquement la méthylation de H3K9, tout comme la méthylation de protéines non histones par les KMTs de H3K9, dans la régulation de la pluripotence et la différenciation cellulaires normales et dans certains contextes pathologiques, tels que le cancer et certaines dystrophies.
Nous étudions les cellules souches musculaires (MuSCs), les cellules souches embryonnaires (ESCs), les cellules pluripotentes induites (iPSCs) et les cellules souches cancéreuses (CSCs) de l’adénocarcinome du poumon. Nous essayons de disséquer la composition, les mécanismes, la cinétique d’action de ces KMTs et le dialogue entre elles. Les résultats attendus vont accroitre nos connaissances sur les mécanismes gouvernant les modifications de la chromatine au cours de la reprogrammation cellulaire, et potentiellement ouvrir la voie vers de nouvelles thérapies ciblées.
Nos approches expérimentales principales : pour étudier les KMTs aussi bien au niveau moléculaire que cellulaire, nous combinons les approches gain et perte de fonction. Nous utilisons CRISPR-based genome editing, des approches épigénomiques (ChIP-seq, bulk and single-cell RNA-seq), protéomiques (TAP-tag/mass spectrometry), bioinformatiques, biophysique du noyau par Atomic Force Microscopy (AFM) et microfluidique (en collaboration), de biologie cellulaire et d’imagerie sophistiquées, wound healing automatisé et de xénogreffes.
Publications
Sélection de publications récentes
-
Epigenetic control of nuclear mechanics and cellular migration via histone H3 lysine 9 methylation at Lamina-Associated Domains. bioRxiv 2025. Silvia Comunian, Valentin Petit, Guillaume Velasco, Vlada Zakharova, Cécile Jebane, Pauline Lahure, Véronique Joliot, Laurence Del Maestro, Magali Hennion, Guillaume Lamour, Jean-Baptiste Manneville, Emmanuèle Helfer, Ekaterina Boyarchuk, Slimane Ait-Si-Ali.
DOI: 11.28.690983 - EMILIN1 emerges as a TGFβ/SETDB1-regulated secreted biomarker in Duchenne Muscular Dystrophy. bioRxiv 2025, Maeva Zamperoni, Laura Muraine, Alice Granados, Anne Bigot, Valentin Petit, Mona Bensalah, Jessica Ohana, Véronique Legros, Ekaterina Boyarchuk, Guillaume Chevreux, Véronique Joliot, Elisa Negroni, Maryline Moulin, Capucine Trollet, Slimane Ait-Si-Ali.
DOI: 10.11.681604 -
Automethylation of lysine methyltransferase SETDB1 on H3K9-like motifs regulates interactions with chromodomain proteins and controls its functions. bioRxiv, 2025. Paola Cruz-Tapias, Guillaume Velasco, Roberta Rapone, Laurence Del Maestro, Victor Cochard, Guillaume Chevreux, Véronique Joliot, Ekaterina Boyarchuk, Slimane Ait-Si-Ali.
DOI: 10.22.683908 - Genetic diseases of epigenetic machinery. Med Sci (Paris), 2024, Maud de Dieuleveult, Guillaume Velasco.
PMID: 39705562 -
Protocol for differential analysis of Trim71-associated protein complexes in mouse embryonic stem cells by mass spectrometry using Perseus. STAR Protoc, 2024, Cosson B, Rapone R, Del Maestro L, Ait-Si-Ali S.
PMID: 39116200 -
Setdb1 protects genome integrity in murine muscle stem cells to allow for regenerative myogenesis and inflammation. Dev Cell, 2024, Garcia P, Jarassier W, Brun C, Giordani L, Agostini F, Kung WH, Peccate C, Ravent J, Fall S, Petit V, Cheung TH, Ait-Si-Ali S, Le Grand F.
PMID: 38848717 - SETDB1 modulates the TGFβ response in Duchenne muscular dystrophy myotubes. Sci Adv, 2024, Granados A, Zamperoni M, Rapone R, Moulin M, Boyarchuk E, Bouyioukos C, Del Maestro L, Joliot V, Negroni E, Mohamed M, Piquet S, Bigot A, Le Grand F, Albini S, Ait-Si-Ali S.
PMID: 38691608 - The cytoplasmic fraction of the histone lysine methyltransferase Setdb1 is essential for embryonic stem cells. iScience, 2023, Rapone R, Del Maestro L, Bouyioukos C, Albini S, Cruz-Tapias P, Joliot V, Cosson B, Ait-Si-Ali S.
PMID: 37559904 - SETDB1 Fuels the Lung Cancer Phenotype by Modulating Epigenome, 3D Genome Organization and Chromatin Mechanical Properties. Nucleic Acids Research, 2022. Zakharova VV, Magnitov MD, Del Maestro L, Ulianov SV, Glentis A, Ulyanik B, Williart A, Karpukhina A, Demidov O, Joliot V, Vassetzky YS, Mège RM, Piel M, Razin SV & Ait-Si-Ali S.
PMID: 35474385 - Glucose controls co-translation of structurally related mRNAs via the mTOR and eIF2 pathways in human pancreatic beta cells. Front Endocrinol. 2022. Bulfoni M, Bouyioukos C, Zakaria A, Nigon F, Rapone R, Del Maestro L, Ait-Si-Ali S, Scharfmann R, Cosson B.
PMID : 35992129 - Paramecium Polycomb repressive complex 2 physically interacts with the small RNA-binding PIWI protein to repress transposable elements. Dev Cell. 2022. Del Maestro L, Ait-Si-Ali S,
PMID : 35429435
- Acute conversion of patient-derived Duchenne muscular dystrophy iPSC into myotubes reveals constitutive and inducible over-activation of TGFβ-dependent pro-fibrotic signaling. Skelet. Muscle 2020. Caputo L, Granados A, Lenzi J, Rosa A, Ait-Si-Ali S, Puri PL, Albini S.
PMID: 32359374 - The H3K9 Methylation Writer SETDB1 and Its Reader MPP8 Cooperate to Silence Satellite DNA Repeats in Mouse Embryonic Stem Cells. Genes, 2019. Cruz-Tapias P, Robin P, Pontis J, Del Maestro L, Ait-Si-Ali S.
PMID: 31557926 - Expression of the Major and Pro-Oncogenic H3K9 Lysine Methyltransferase SETDB1 in Non-Small Cell Lung Cancer. Cancers, 2019. Cruz-Tapias P, Zakharova V, Perez-Fernandez OM, Mantilla W, Ramírez-Clavijo S, Ait-Si-Ali S.
PMID: 31398867 - Canonical Wnt signalling regulates nuclear export of Setdb1 during skeletal muscle terminal differentiation. Cell Discovery, 2016. S Beyer, J Pontis, E Schirwis, V Battisti, A Rudolf, F Le Grand, S Ait-Si-Ali.
PMID: 27867535 - Unexpected antagonistic roles of the related histone H3 lysine 9 methyltarsferase G9a and G9a-Like Protein in proliferating but not in differentiating myoblasts. J Mol Biol, 2016. V Battisti, J Pontis, Boyarchuk E, Fritsch L, P Robin, S Ait-Si-Ali and V Joliot.
PMID: 27056598 - Sound of Silence: the properties and functions of repressive lysine methyltransferases. Nature Reviews Molecular Cell Biology, 2015. Mozzetta C, Boyarchuk E, Pontis J and Ait-Si-Ali S.
PMID: 26204160
Contact
CNRS – Epigenetics and Cell Fate – UMR7216
Université Paris Cité
Bâ Lamarck, 4ème étage. Case courrier 7042
35, rue Hélène BRION
75205 Paris Cedex 13
Tél : 33 (0)1 5727 8919
Fax : 33 (0)1 5727 8911
Email : slimane.aitsiali@u-paris.fr
Membres
Financements
À lire aussi

Bienvenue à Léa
Léa rejoint l’équipe en tant qu’ingénieure d’études. Après un master en virologie, elle a travaillé à Strasbourg sur les virus de la vigne, puis sur la caractérisation de la dégradation des ARNm chez les plantes à l’Institut de biologie moléculaire des plantes (IBMP)....

Sophie Polo reçoit un financement Impulscience® de la Fondation Bettencourt Schueller
Sophie Polo a reçu un financement Impulscience® pour financer un projet de recherche sur l'établissement et le maintien du chromosome X inactif en réponse aux cassures de l'ADN. C'est une formidable nouvelle pour le laboratoire! Nous remercions la Fondation...
Quand retirer une étiquette de l’ADN stoppe la croissance tumorale
Chez les mammifères, la méthylation de l’ADN est une petite modification chimique qui agit comme une étiquette sur certaines bases de l’ADN, les cytosines. Elle n’altère pas la séquence mais participe au contrôle de l’activité des gènes et la stabilité du génome. Dans...

Bienvenue à Léa, nouvelle ingénieure d’études dans l’équipe!
Léa rejoint l'équipe en tant qu'ingénieure d'études. Titulaire d'un Master en biologie moléculaire et cellulaire de Sorbonne Université, Léa va contribuer à un projet de recherche visant à disséquer les mécanismes de maintien de la méthylation de l'ADN en réponse aux...