Équipe Polo
Intégrité de l’épigénome
L’équipe de Sophie Polo étudie la plasticité de la chromatine en réponse aux dommages de l’ADN dans les cellules de mammifères. Elle cherche à identifier les facteurs qui contrôlent la dynamique des histones, des marques de chromatine et plus généralement les altérations de la structure et de la fonction de la chromatine en réponse au stress génotoxique. Réciproquement, l’équipe étudie l’impact des altérations de l’épigénome sur la stabilité du génome en contexte physiologique et pathologique. En combinant des approches de protéomique avec des techniques d’imagerie de pointe, les travaux de l’équipe visent à comprendre comment le maintien de l’intégrité du génome et l’épigénome sont coordonnés.
Equipe Intégrité de l’épigénome
(Avril 2025)
De gauche à droite:
Debout: Sophie Polo, Annabelle Shaw, Audrey Chansard, Julia Roche Dupuy, Giulia Giacomini, Delphine Burlet.
Devant: Sandra Piquet, Juliette Dabin, Margherita Mori, Eliane Petit.
Dans ce contexte, nous cherchons à comprendre comment l’information véhiculée par la chromatine est préservée lorsque son intégrité est mise en péril par un stress génotoxique et comment le maintien de l’intégrité du génome et de l’épigénome sont coordonnés en conditions physiologiques et lors du dévelopement tumoral. Pour cela, nous étudions les altérations induites par les dommages à l’ADN dans des cellules mammifères à différents niveaux d’organisation de la chromatine, des protéines histones aux niveaux de compaction supérieurs de la chromatine, et nous explorons les mécanismes sous-jacents. Réciproquement, nous analysons les conséquences des altérations de la chromatine, incluant les oncomutations d’histones, sur le maintien de l’intégrité du génome.
Nos approches expérimentales combinent la biologie moléculaire et la protéomique avec des techniques d’imagerie de pointe pour étudier la dynamique de la chromatine en réponse au stress génotoxique dans des cellules de mammifères en culture. Elles incluent:
- l’induction locale de dommages à l’ADN par micro-irradiation ou la technologie CRISPR/Cas9,
- le marquage in vivo des protéines histones à l’aide de la technologie SNAP-tag,
- le suivi dynamique des marques épigénétiques – modifications de l’ADN et des histones – dans les zones de réparation de l’ADN.
Nos projets de recherche:
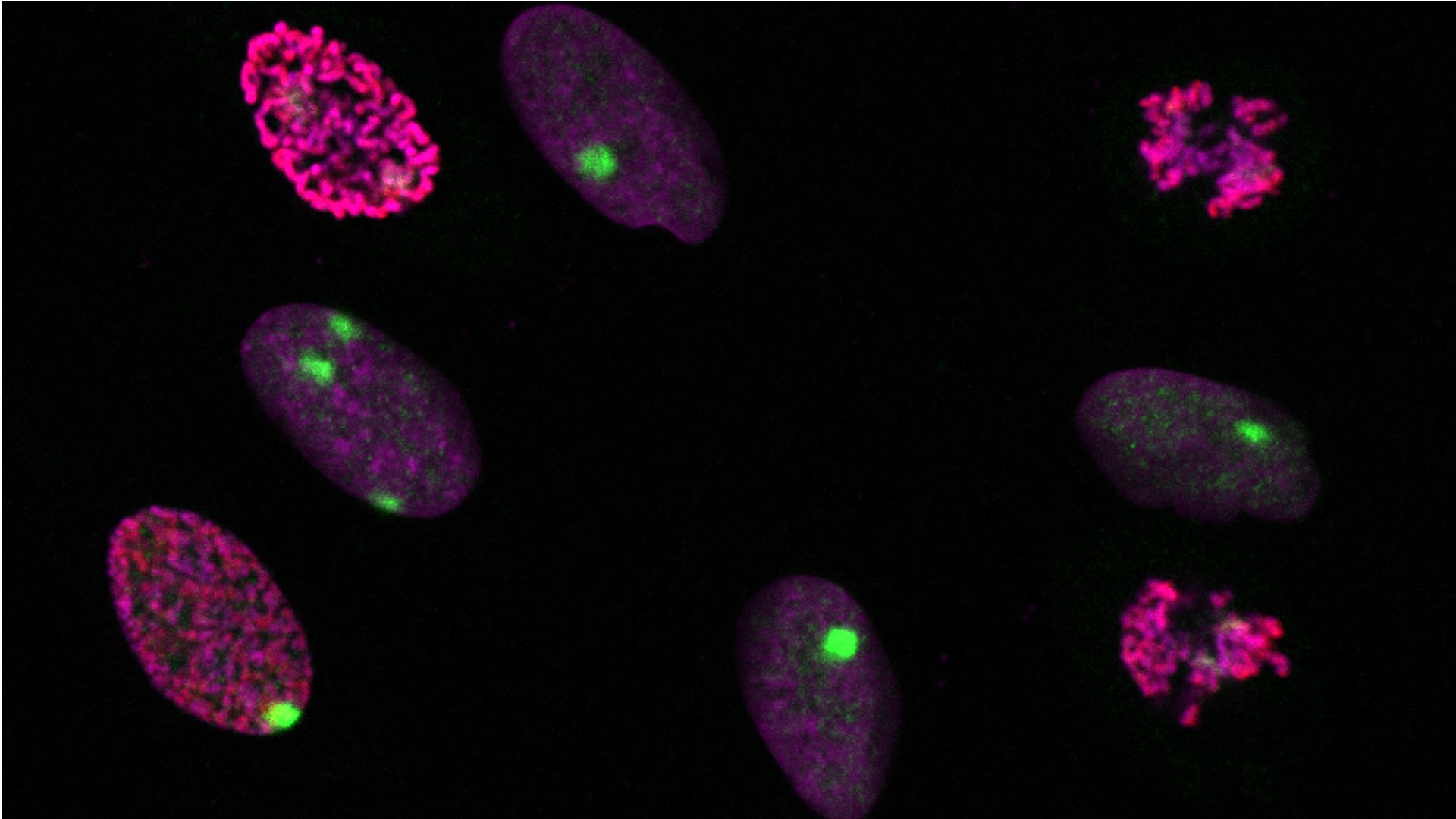
Dynamique des modifications de l’ADN et des histones aux sites de réparation
Description
Afin de déterminer comment les états épigénétiques sont transmis lors de la réparation de l’ADN, nous profilons l’épigénome des zones de réparation des dommages UV dans les cellules de mammifères, en nous concentrant sur les modifications post-traductionnelles des histones et la méthylation de l’ADN.
Pour caractériser de façon exhaustive et non biaisée les changements dynamiques des modifications d’histones pendant la réparation des lésions UV, nous avons conçu deux approches protéomiques complémentaires basées sur (1) la biotinylation des protéines aux sites de réparation et sur (2) la capture des sites de synthèse réparatrice, combinées à des analyses de spectrométrie de masse. En parallèle, nous avons adopté une approche candidate centrée sur les phosphorylations mitotiques d’histones aux sites de dommages UV par imagerie. Nous menons ensuite des études fonctionnelles pour disséquer les mécanismes qui sous-tendent les altérations des modifications d’histones pendant la réparation des dommages causés par les UV et leurs conséquences sur le destin cellulaire.
Pour étudier le maintien de la méthylation de l’ADN aux sites de réparation des dommages UV, nous combinons des approches d’imagerie et de séquençage à haut débit sur l’ADN en cours de synthèse réparatrice. Nous cherchons à identifier les acteurs moléculaires impliqués, et les connections potentielles avec la dynamique des histones.
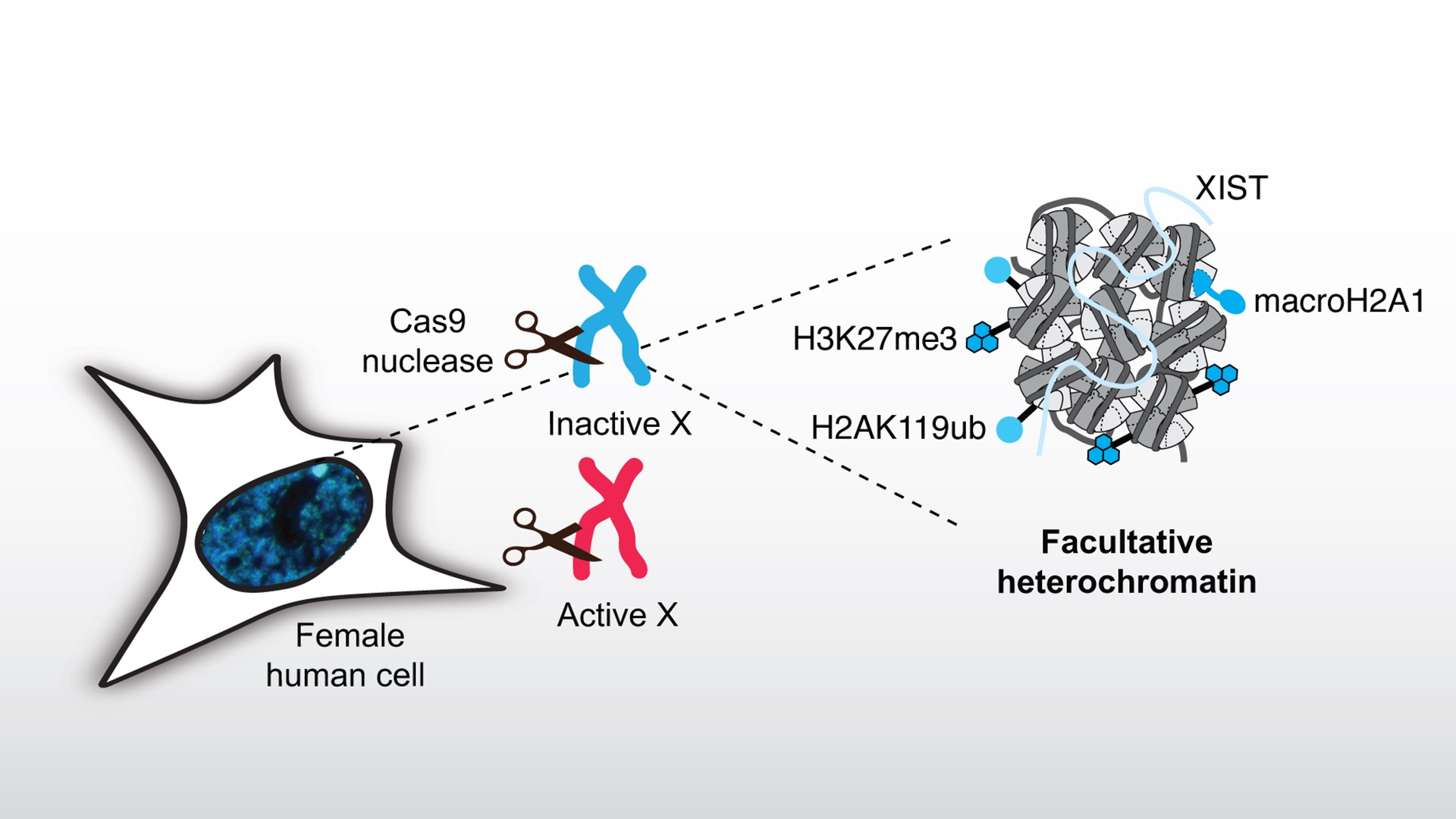
Réponse aux dommages à l’ADN dans les domaines d’hétérochromatine facultative
Description
Les domaines compacts d’hétérochromatine représentent un défi pour la réparation des dommages à l’ADN, comme en attestent leurs taux élevés de mutations. Pour étudier la réponse de l’hétérochromatine facultative aux dommages à l’ADN, nous nous focalisons sur le chromosome X inactif (Xi) qui est soumis à une inactivation transcriptionnelle par la formation d’hétérochromatine facultative pendant le développement embryonnaire chez les mammifères femelles.
Nous induisons des cassures double brin (DSBs) de l’ADN avec la nucléase Cas9 dans les chromosomes X et examinons comment le paysage de l’hétérochromatine facultative dicte le choix de la voie de réparation des cassures. Nous testons également l’hypothèse selon laquelle l’induction de DSBs peut guider le choix du chromosome X à inactiver dans les cellules souches embryonnaires. Réciproquement, nous analysons l’impact de la réparation des DSBs sur le maintien ou l’altération des marques chromatiniennes du Xi et nous explorons leur impact fonctionnel sur l’inactivation transcriptionnelle des gènes liés au chromosome X. En particulier, nous nous concentrons sur les altérations de la marque d’hétérochromatine facultative H3K27me3 en réponse aux DSBs et sur la pertinence fonctionnelle d’une régulation fine de cette marque d’histone pour la stabilité du génome.
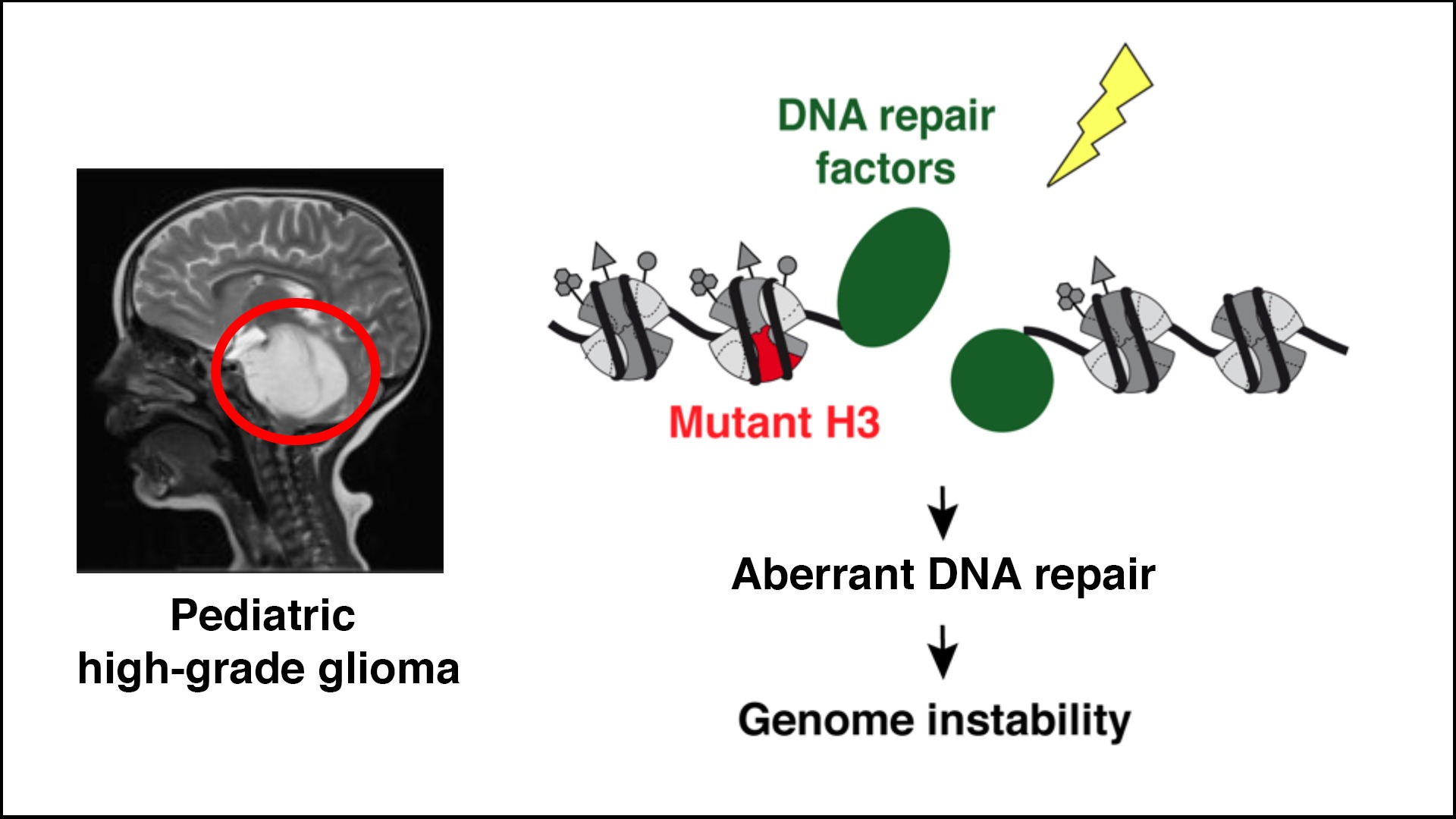
Impact des oncomutations d’histones sur le maintien de l’intégrité du génome
Description
Les mutations ponctuelles des variants de l’histone H3 sont directement impliquées dans plusieurs types de cancers, dont le gliome pédiatrique de haut grade, une maladie mortelle et actuellement incurable. Afin d’ouvrir de nouvelles pistes thérapeutiques, nous testons l’hypothèse selon laquelle les mutations associées aux gliomes dans les histones H3 (K27M, G34R/V) peuvent rendre le génome instable en altérant la réparation de l’ADN. Une réparation aberrante de l’ADN constitue une vulnérabilité qui peut être ciblée pour contrer la croissance des cellules cancéreuses.
Nous combinons des approches génomiques, protéomiques et d’imagerie dans des tumeurs et des modèles cellulaires avec des études précliniques in vivo pour (1) disséquer et exploiter le dysfonctionnement des enzymes de réparation de l’ADN dans les cellules mutantes sur l’histone H3, (2) caractériser les mécanismes chromatiniens par lesquels les histones mutantes conduisent à la réparation aberrante de l’ADN, et (3) comparer la réponse aux dommages de l’ADN résultant des oncomutations sur H3.1 et H3.3.
Publications
2025
- Ferrand J.*, Dabin J.*, Chevallier O., Kane-Charvin M., Kupai A., Hrit J., Rothbart S., and Polo S.E. Mitotic chromatin marking drives the segregation of DNA damage. Nat Commun, 16: 746, 2025. *: equal contribution
2024
- Dabin J.*, Giacomini G.*, Petit E.*, and Polo S.E. New facets in the chromatin-based regulation of genome maintenance. DNA Repair, 140: 103702, 2024. *: equal contribution
- Saredi G., Carelli F.N., Rolland S.*, Furlan G.*, Piquet S.*, Appert A.*, Sanchez-Pulido L., Price J.L., Alcon P., Lampersberger L., Déclais A-C., Ramakrishna N.B., Toth R., Macartney T., Alabert C., Ponting C.P., Polo S.E., Miska E.A., Gartner A., Ahringer J., and Rouse J. The histone binding capacity of SPT2 controls chromatin structure and function in Metazoa. Nat Struct Mol Biol, 2024, doi : 10.1038/s41594-023-01204-3. *: equal contribution
- Giacomini G., Piquet S., Chevallier O., Dabin J., Bai S-K., Kim B., Siddaway R., Raught B., Coyaud E., Shan C-M., Reid R.J.D., Toda T., Rothstein R., Barra V., Wilhelm T., Hamadat S., Bertin C., Crane A., Dubois F., Bandopadhayay P., Beroukhim R., Naim V., Jia S., Hawkins C., Rondinelli B.* and Polo S.E.* Aberrant DNA repair reveals a vulnerability in histone H3.3-mutant brain tumors. Nucleic Acids Res, 2024. doi: 10.1093/nar/gkad1257. *: co-corresponding authors
2023
- Dabin J.*, Mori M.*, Polo S.E. The DNA damage response in the chromatin context: a coordinated process. Current Opin Cell Biol, 82: 102176, 2023. *: equal contribution
2022
- Chansard A.*, Pobega E.*, Caron P., Polo S.E. Imaging the response to DNA damage in heterochromatin domains. Front Cell Dev Biol, 10: 920267, 2022. *: equal contribution
2021
- Caron P.*, Pobega E.*, Polo S.E. DNA double-strand break repair: All roads lead to heterochROMAtin marks. Front Genet, 12: 730696, 2021. *: equal contribution
- Bouvier D., Ferrand J., Chevallier O., Paulsen M.T., Ljungman M., Polo SE. Dissecting regulatory pathways for transcription recovery following DNA damage reveals a non-canonical function of the histone chaperone HIRA. Nat Commun, 12: 3835, 2021.
- Olley G., Madapura P., Grimes G., Piquet S., Polo S.E., FitzPatrick D., Bickmore W., Boumendil C. Cornelia-de Lange syndrome-associated mutations cause a DNA damage signalling and repair defect. Nat Commun, 12: 3127, 2021.
- Caron P., Pobega E., Polo S.E. A molecular Rosetta Stone to decipher the impact of chromatin features on the repair of Cas9-mediated DNA double-strand breaks. Mol Cell, 81:2059-2060, 2021.
- Fortuny A., Chansard A., Caron P., Chevallier O., Leroy O., Renaud O., Polo S.E. Imaging the response to DNA damage in heterochromatin domains reveals core principles of heterochromatin maintenance. Nat Commun, 12: 2428, 2021.
- Ferrand J.*, Plessier A.*, Polo S.E. Control of the chromatin response to DNA damage: Histone proteins pull the strings. Semin Cell Dev Biol, 113:75-87, 2021. *: equal contribution
2020
- Ferrand J.*, Rondinelli B.*, Polo S.E. Histone variants: guardians of genome integrity. Cells, 9:2424, 2020. *: equal contribution
- Ahmad R, Lahuna O, Sidibe A, Daulat A, Zhang Q, Luka M, Guillaume JL, Gallet S, Guillonneau F, Hamroune J, Polo S, Prévot V, Delagrange P, Dam J, Jockers R. GPR50-Ctail cleavage and nuclear translocation: a new signal transduction mode for G protein-coupled receptors. Cell Mol Life Sci, 77: 5189-5205, 2020.
- Caron P., Polo S.E. Reshaping Chromatin Architecture around DNA Breaks. Trends Biochem Sci, 45: 177-179, 2020.
2018
- Piquet S., Le Parc F., Bai, S-K., Chevallier O., Adam S., Polo S.E. The histone chaperone FACT coordinates H2A.X-dependent signaling and repair of DNA damage. Mol Cell, 72: 888-901, 2018.
- Dabin J.*, Fortuny A.*, Piquet S, Polo S.E. Live imaging of parental histone variant dynamics in UVC-damaged chromatin. Methods Mol Biol, 1832: 243-253, 2018. *: equal contribution
- Fortuny A, Polo S.E. The response to DNA damage in heterochromatin domains. Chromosoma, 127: 291-300, 2018.
2017
- Fortuny A., Polo S.E. Genome and epigenome maintenance by keeping histone turnover in check. Mol Cell, 66: 3-4, 2017
- Dabin J., Polo S. E. Choreography of parental histones in damaged chromatin. Nucleus, 8: 255-260, 2017
- Polo S.E. Switching genes to silent mode near DNA double-strand breaks. EMBO Rep, 18:659-660, 2017.
2016
- Adam S.*, Dabin J.*, Chevallier O., Leroy O., Baldeyron C., Corpet A., Lomonte P., Renaud O., Almouzni G. and Polo S.E. Real-time tracking of parental histones reveals their contribution to chromatin integrity following DNA damage. Mol Cell, 64: 65-78, 2016. *: equal contribution. Featured article, also highlighted in the « Meet the author » section
- Dabin J.*, Fortuny A.* and Polo S.E. Epigenome maintenance in response to DNA damage. Mol Cell, 62: 712-727, 2016. *: equal contribution
2015
- Polo S.E.*, Almouzni G.* Chromatin plasticity in response to DNA damage: the legacy of the access-repair-restore model. DNA Repair, 36: 114-121, 2015. *: co-corresponding author
- Adam S.*, Dabin J.* and Polo S.E. Chromatin plasticity in response to DNA damage: the shape of things to come. DNA Repair, 32:120-126, 2015. *: equal contributio
- Adam S.*, Dabin J.*, Bai S-K. and Polo S.E. Imaging local deposition of newly synthesized histones in UVC-damaged chromatin. Methods Mol Biol, 1288-337-347, 2015. *: equal contributio
- Polo S.E. Reshaping chromatin after DNA damage: the choreography of histone proteins. J Mol Biol, 427:626-636, 2015.
2014
- Adam S., Polo S.E. Blurring the line between the DNA damage response and transcription: the importance of chromatin dynamics. Exp Cell Res, 329:148-153, 201
- Adam S., Polo S. E. and Almouzni G. How to restore chromatin structure and function in response to DNA damage – let the chaperones play. FEBS J, 281: 2315-2323, 2014
2013
- Adam S., Polo S. E.* and Almouzni G.* Transcription recovery after DNA damage requires chromatin priming by the H3.3 histone chaperone HIRA. Cell, 155:94-106, 2013. *: co-corresponding author
- Polo S. E. Fine-tuning the p53 response to DNA damage: a new piece in the puzzle. Cell Cycle, 12: 1337-1338, 2013.
Membres

Delphine Burlet
Post-doctoral fellow (INCa)
delphine.burlet-thomas@cnrs.fr
+33 1 57 27 89 32
READ MORE
Audrey Chansard
Research assistant | EPI2 Imaging Platform (CNRS)
audrey.chansard@univ-paris-diderot.fr
+33 1 57 27 89 81
READ MORE
Juliette Dabin
Post-doctoral fellow (MSCA)
juliette.dabin@univ-paris-diderot.fr
+33 1 57 27 89 81
READ MORE
Margherita Mori
PhD student (Univ. Paris Cité)
margherita.mori@etu.u-paris.fr
+33 1 57 27 89 32
READ MORE
Sandra Piquet
Research assistant & lab manager | EPI2 Imaging Platform manager (CNRS)
+33 1 57 27 89 81
READ MORE-
Anciens membres
Salomé Adam
Etudiante en thèse (Sept 2011-Juin 2015)
Postdoc (Juin 2015-Sept 2015)
Après un stage postdoctoral très fructueux dans le laboratoire de Dan Durocher à Toronto (2015-2021), Salomé a rejoint AstraZeneca, Cambridge, GB en tant que chargée de recherche senior.
Siau-Kun Bai
Lab manager (Jan 2013-Mar 2014)
Siau est ingénieure d’études dans le laboratoire de Ludger Johnannes à l’Institut Curie, Paris.
Ines Beckerman
Etudiante invitée (Sept-Dec 2024)
Ines est retournée finir sa thèse dans le laboratoire du Dr. Manuel Muñoz à l’Université de Buenos Aires en Argentine.
Zoé Begué
Etudiante en Master 1 (Avr-Juin 2022)
Zoé a repris ses études de Master à l’Université Paris-Saclay.
Camille Boucher
Ingénieure d’études (Juil 2022-Juil 2023) dans le cadre du programme transverse Labex sur les organoïdes intestinaux
Camille est désormais ingénieure d’études dans l’équipe de Béatrice Romagnolo à l’Institut Cochin, Paris
Déborah Bouvier
Ingénieure d’études (Jan 2016-Sept 2020)
Déborah est désormais attachée de recherche clinique chez IQVIA
Jeanne Brouillet
Etudiante en Master 1 (Juin-Août 2021)
Jeanne a repris ses études de Master à AgroParisTech
Nolan Caile
Stagiaire (Fev-Avr 2024)
Nolan a repris ses études à Tufts University, Boston, USA
Pierre Caron
Post-doc (Avr 2019-Dec 2021)
Après un autre stage postdoctoral dans le laboratoire de Beatrice Eymin à l’IAB de Grenoble, Pierre est désormais chargé de recherche CNRS dans le groupe de Joanna Timmins à l’Institut de Biologie Structurale de Grenoble
Odile Chevallier
Ingénieure d’études à mi-temps (Jan 2014-Fev 2023)
Après 9 ans de travail assidu dans l’équipe aboutissant à 4 articles de recherche publiés et 2 en cours, Odile profite désormais d’une retraite bien méritée.
Arnaud Debernardi
Etudiant en Master 1 (Jan-Fev 2020)
Arnaud a repris ses études de Master à l’Université de Lyon
Marc El Hasbany
Etudiant en Master 1 (Avr-Juil 2024)
Après son stage utilisant les simulations par dynamique moléculaire co-supervisé par Leslie Regad (Univ. Paris Cité), Marc a repris ses études de Master à l’Université Paris Cité.
Juliette Ferrand
Etudiante en Master 2 (Jan 2017-Juin 2017)
Etudiante en thèse (Sept 2017-Sept 2021)
Post-doc (Sept 2021-Dec 2022)
Juliette a rejoint le laboratoire de Niels Mailand à Copenhague pour un stage postdoctoral.
Anna Fortuny
Etudiante en Master 2 (Jan 2015-Juin 2015)
Etudiante en thèse (Sept 2015-Sept 2019)
Post-doc (Sept 2019-Dec 2020)
Anna est retournée en Catalogne pour travailler dans le départmement R&D d’une compagnie de biotechnologies spécialisée dans les tests immunologiques
Philippine Harou
Etudiante en 3e année de licence (Nov-Dec 2023)
Philippine a repris ses études en licence à l’Université Paris Cité.
Matteo Kané-Charvin
Etudiant en 2e année de médecine (Juin-Sept 2022)
Matteo a repris ses études de médecine à l’Université Paris Cité.
Florent Le Parc
Lab manager (Mar 2014-Dec 2015)
Florent a quitté la recherche académique pour suivre une formation de soigneur animalier.
François Martin
Stagiaire (Juin-Août 2023)
François a repris ses études d’ingénieur à Polytech Nice-Sophia.
Alexandre Plessier
Post-doc (Avr 2019-Sept 2023)
Alex est désormais manager de projet à l’Université Paris Cité
Enrico Pobega
Etudiant en thèse (Sept 2019-Dec 2023)
Enrico est retourné à l’Université de Trieste en Italie pour un stage postdoctoral.
Beatrice Rondinelli
Postdoc Marie Curie (Sept 2017- Sept 2018)
Chargée de recherche CNRS (Oct 2018-Avr 2022)
Beatrice a obtenu un financement ATIP-avenir en 2021 lui permettant de monter sa propre équipe de recherche en Mai 2022 à l’Institut Gustave Roussy
Hicham Ziaina
Etudiant en Master 1 (Janv-Févr 2024)
Hicham a repris ses études de Master à l’Université Claude Bernard de Lyon.
Contact
Sophie Polo, PhD
Centre Epigénétique et Destin Cellulaire
CNRS UMR7216 – Université Paris Cité
Bâtiment Lamarck B, 4e étage, pièce 413
35, rue Hélène Brion
75205 Paris Cedex 13
Tel : 33 (0)1 57 27 89 81
Fax : 33 (0)1 57 27 89 11
Email : sophie.polo@u-paris.fr
Nous recherchons toujours des personnes talentueuses et motivées pour rejoindre notre équipe.
N’hésitez pas à nous contacter si vous êtes intéressés.
À lire aussi

Félicitations à Delphine et Julia pour le financement de leurs projets de recherche
Félicitations à Delphine Burlet pour l'obtention d'un financement postdoctoral de la Fondation de France et à Julia Roche Dupuy pour avoir décroché une bourse de l'école doctorale Hématologie-Oncogénèse-Biothérapies pour financer sa thèse. Delphine Burlet, Julia Roche...

Bienvenue à Annabelle, nouvelle post-doctorante dans l’équipe!
Annabelle rejoint le laboratoire en tant que post-doctorante. Elle a réalisé sa thèse à l'Université d'Oxford sous la direction de Monika Gullerova. Ses travaux de doctorat ont porté sur le rôle des petits ARNs non codants de type Y et de la protéine qui les lie,...

Bienvenue à Delphine, nouvelle post-doctorante dans l’équipe!
Delphine rejoint le laboratoire en tant que post-doctorante. Elle a réalisé sa thèse au Centre de Recherche en Cancérologie de Lyon (CRCL) sous la direction de Virginie Petrilli. Ses travaux de doctorat ont porté sur la protéine pro-inflammatoire NLRP3, dont elle a...
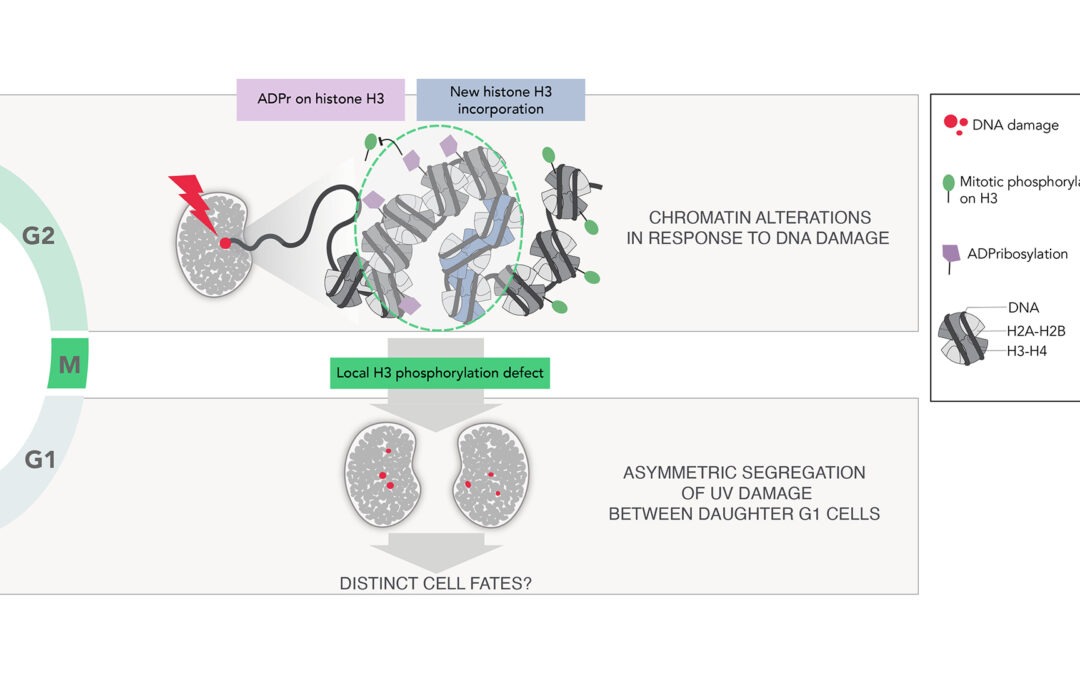
Nouvel article de recherche sur le contrôle de la ségrégation des dommages à l’ADN par des altérations de la chromatine
Dans cette étude, nous avons découvert un système de marquage de la chromatine endommagée par les UV qui gouverne la ségrégation des dommages à l’ADN au cours de la mitose, avec des conséquences potentielles sur le destin des cellules filles. Ainsi, nous montrons que...


















