Équipe Rougeulle
ARN non codants, Différentiation et Développement

© Equipe Rougeulle
La recherche dans le laboratoire de Claire Rougeulle se concentre sur le processus emblématique de l’inactivation du chromosome X, dans le but d’élucider comment une telle extinction épigénétique à l’échelle du chromosome est réalisée et régulée dans diverses espèces de mammifères. Plus généralement, nous nous intéressons au contrôle épigénétique de l’expression des gènes en relation avec l’identité et le destin des cellules, à la contribution des gènes générant des longs ARNs non codant et des éléments transposables à ces processus et à la plasticité des régulations épigénétiques dans l’évolution.
Nous utilisons une variété de cellules souches pluripotentes de souris et de primates et leurs dérivés différenciés pour récapituler les étapes clés du développement et les états cellulaires. Notre stratégie expérimentale combine l’ingénierie des génomes (CRISPR) avec des analyses de cellules uniques, des enquêtes transcriptomiques et épigénomiques à grande échelle et des études topologiques en 3D.
Nos projets de recherche:
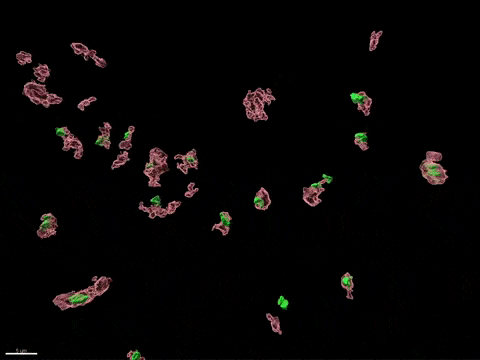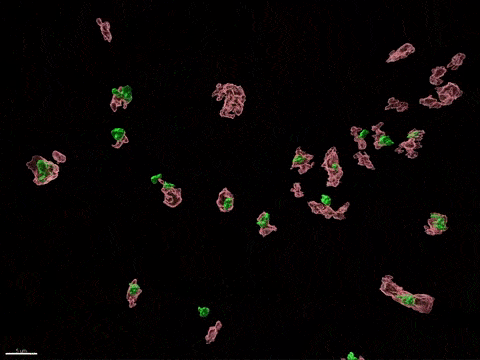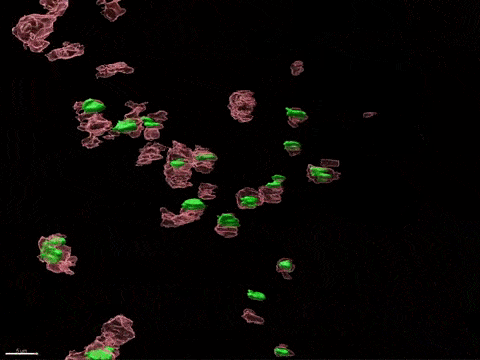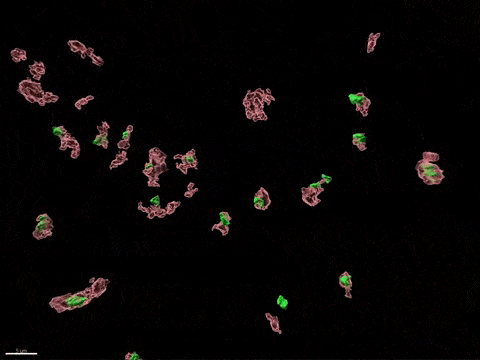
Inactivation du chromosome X humain in vitro
Description
L’inactivation du chromosome X (XCI) est essentielle au développement de la femme et liée à des susceptibilités spécifiques au sexe pour diverses maladies. Pour comprendre comment l’inactivation du chromosome X contribue aux différences entre les hommes et les femmes, nous modélisons, in vitro, les étapes clés de l’inactivation du chromosome X en utilisant, comme point d’entrée, les cellules souches embryonnaires humaines (CSEh). Les CSEh sont reprogrammées pour découvrir comment l’inactivation du chromosome X est initialement établie au cours des premiers jours du développement embryonnaire, avec un accent particulier sur le rôle des ARN non codants structurels tels que XIST et XACT. Les CSEh sont différenciés en types de cellules spécifiques pour évaluer la plasticité de XCI dans des tissus somatiques distincts, y compris le compartiment hématopoïétique et le tissu organisé par l’utilisation d’organoïdes.

Évolution de l’inactivation du chromosome X chez les primates
Description
Il existe des différences remarquables dans la manière dont l’inactivation du chromosome X est mise en place entre l’homme et la souris, en relation avec un réseau radicalement différent de gènes lncRNA régulateurs que notre groupe a contribué à déchiffrer. Nous explorons maintenant la variabilité phénotypique et mécanistique de l’inactivation du chromosome X chez les primates en utilisant des cellules souches pluripotentes de primates non humains afin de sonder le degré d’innovation génétique et épigénétique sur une échelle de temps limitée, et la contribution du génome non codant à cette évolution.
Plasticité du génome non codant
Description
Le génome non codant participe à l’établissement des programmes d’expression des gènes, et, à ce titre, à l’identité et au destin des cellules. Il englobe un vaste répertoire de gènes lncRNA (LRGs) – dont une fraction provient de rétrovirus endogènes – dont la fonction peut être véhiculée par des modules distincts, tels que l’ARN lui-même, le locus génomique, l’acte de transcription ou tout sous-produit ARN plus petit en aval. L’inactivation du X est un exemple frappant d’un processus contrôlé par les GRL agissant à plusieurs niveaux et par divers mécanismes. Notre objectif est d’explorer la polyvalence des LRGs afin de comprendre si un LRG donné peut jouer de multiples fonctions dans divers contextes développementaux et cellulaires, et comment les mécanismes sous-jacents peuvent varier en conséquence.
Publications
2024
- Morey C., Rougeulle C. & Ouimette JF. Unleashing XIST from X-chromosome inactivation. Current Opinion in Cell Biology, 2024 November
- Alfeghaly C., Castel G., Cazottes E., Moscatelli M., Moinard E., Casanova M., Boni J., Mahadik K., Lammers J., Freour T. , Chauviere L.,Piqueras C., Boers R., Boers J., Gribnau J.,David L., Ouimette JF. & Rougeulle C. XIST dampens X chromosome activity in a SPEN-dependent manner during early human development. Natural Structural and Molecular Biology, 2024 June
- Altered X-chromosome inactivation predisposes to autoimmunity . Science Advances, 2024 May
2023
- Rosspopoff O, Cazottes E, Huret C, Loda A, Collier A, Casanova M, Rugg-Gunn P, Heard E, Ouimette JF, Rougeulle C. Species-specific regulation of XIST by the JPX/FTX orthologs . Nucleic Acids Research, 2023 Feb
2022
- Mahadik K, Rougeulle C. Study of X Chromosome Activity Status in Human Naive Pluripotent Stem Cells Using RNA-FISH Methods Mol Biol. 2022;2416:239-255
2021
- Heard E, Rougeulle C. Digging into X chromosome inactivation. Science. 2021 Nov 19;374(6570):942-943.
- Moscatelli M., Rougeulle C. Dernières nouvelles du chromosome X: des principes généraux nuancés. Médecine/Sciences, 37 2 (2021) 152-158
2020
- Ouimette JF, Rougeulle C. Many XCI-ting routes to reach the eXACT dose. Nat Cell Biol. 2020 Dec;22(12):1397-1398
- Cazottes E., Rougeulle C. Straight to the X: Modeling Human X Chromosome Inactivation in hESCs by FGF Signal Blockade.Cell Stem Cell. 2020 Sep 3;27(3):352-353
- Patrat C, Ouimette JF, Rougeulle C. X chromosome inactivation in human development. Development 2020 Jan 3;147(1).
2019
- Casanova M, Moscatelli M, Chauvière LÉ, Huret C, Samson J, Liyakat Ali TM, Rosspopoff O, Rougeulle C. A primate-specific retroviral enhancer wires the XACT lncRNA into the core pluripotency network in humans. Nat Commun. 2019 Dec 11;10(1):5652.
2018
- Furlan G, Gutierrez Hernandez N, Huret C, Galupa R, van Bemmel JG, Romito A, Heard E, Morey C, Rougeulle C. The Ftx Noncoding Locus Controls X Chromosome Inactivation Independently of Its RNA Products. Mol Cell. 2018 Apr 20. pii: S1097-2765(18)30227-2.
2017
- Vallot C., Patrat C., Collier AJ., Huret C., Casanova M., Liyakat Ali TM., Tosolini M., Frydman N., Heard E., Rugg-Gunn P.J.and Rougeulle C. Competing action of XIST and XACT noncoding RNAs in the control of X chromosome activity during human early development. Cell Stem Cell. 2017 Jan 5;20(1):102-111.
2016
- Vallot C., Ouimette JF., Rougeulle C. Establishment of X chromosome inactivation and epigenomic features of the inactive X depend on cellular contexts. Bioessays. 2016 Sep;38(9):869-80
- Ouimette JF, Rougeulle C. How Many Non-coding RNAs Does It Take to Compensate Male/Female Genetic Imbalance? Adv Exp Med Biol. 2016;886:33-49
2015
- Vallot C*, Ouimette JF*, Makhlouf M, Féraud O, Pontis J, Côme J, Martinat C, Bennaceur-Griscelli A, Lalande M, Rougeulle C. Erosion of X Chromosome Inactivation in Human Pluripotent Cells Initiates with XACT Coating and Depends on a Specific Heterochromatin Landscape.Cell Stem Cell. 2015 May 7;16(5):533-46.
2014
- Makhlouf M.*, Ouimette JF.*, Oldfield A.*, Navarro P., Neuillet D. and Rougeulle C. A prominent and conserved role for YY1 in Xist transcriptional activation. Nat Commun. 2014 Sep 11;5:4878.
2013
- Häfner S. and Rougeulle C. La matière noire du génome. Pour la Science, 81: 58-63.
- Vallot C., Huret C., Lesecque Y., Resch A., Oudrhiri N., Bennaceur-Griscelli A., Duret L. and Rougeulle C. XACT, a long non-coding transcript coating the active X chromosome in human pluripotent cells. Nat Genet. 2013 Mar;45(3):239-41.
2012
- Vallot C. and Rougeulle C. Epigenetic stability of human pluripotent stem cells. « Epigenomics: From Chromatin Biology to Therapeutics » 118-133.
2011
- Romito A. and Rougeulle C. Origin and evolution of the long non-coding genes in the X-inactivation center. Biochimie. 2011 Nov;93(11):1935-42.
- Makhlouf M. and Rougeulle C. Linking X Chromosome Inactivation to Pluripotency: Necessity or Fate? Trends Mol Med. 2011 Jun;17(6):329-36.
2010
- Navarro P., Oldfield A., Legoupi J., Festuccia N., Dubois A., Attia M., Schoorlemmer J., Rougeulle C., Chambers I. and Avner P. Molecular coupling of Tsix regulation and pluripotency. Nature 2010 Nov 18;468(7322):457-60.
- Mitjavila-Garcia M., Bonnet M.L., Yates F., Haddad R., Oudrhiri N., Féraud O., Magniez A., Vallot C., Makhlouf M., Rougeulle C., Bennaceur-Griscelli A. and Turhan A.G. Partial reversal of the methylation pattern of X-linked gene HUMARA during hematopoietic differentiation of human embryonic stem cells. J Mol Cell Biol. 2010 Oct;2(5):291-8.
2009
- Navarro P., Chantalat S., Foglio M., Chureau C., Vigneau S., Clerc P. Avner P. and Rougeulle C. A role for non-coding Tsix transcription in partitioning chromatin domains within the mouse X-inactivation centre. Epigenetics Chromatin. 2009 Jul 20;2(1):8.
Membres

Jeanne Brouillet
Bioinformatic engineer (CDD CNRS ERC XCycle)
brouillet.jeanne@parisepigenetics.com
+33(0)157278930
READ MORE
Romina Facchinello
PhD student (Université Paris Cité)
romina.facchinello@etu.u-paris.fr
+33 (0)157278930
READ MORE
Christophe Huret
Lab Manager | Vectorology platform manager (Université Paris Cité)
+33(0)157278929
READ MORE
Céline Morey
Senior researcher (INSERM)
celine.morey@univ-paris-diderot.fr
+33(0)157278929
READ MORE
Jean-Francois Ouimette
Assistant Professor (Université Paris Cité)
jean-francois.ouimette@u-paris.fr
+33(0)157278929
READ MOREAnciens membres
Megha Balachandra – Étudiante Master 2
Naïs Boistol – Étudiante BTS
Juliette Boni – Étudiante Master 1
Charlie Bouchu – Étudiant Master 1
Syrlène Bouabdallah – Étudiante Master 2
Léo Carrillo – Etudiant PhD
Miguel Casanova – Post-Doc
Gaël Castel – Ingénieure Bionformatique
Emmanuel Cazottes – Etudiant PhD / Post Doc
Louis Chauvière – Étudiant PhD
Solène Coste – Étudiante Master 2
Arnaud Divol – Étudiant Master 2
Giulia Furlan – Etudiante PhD / Post Doc
Angélique Galvani – Maitre de Conference
Elena Gomez Garcia – Étudiante Licence 2 Erasmus
Nancy Gutierrez Hernandez – Étudiante Master 1
Sophia Häfner – Étudiante PhD
Margot Hully – Étudiante Master 1 ENS
Yoann Kovacs – Étudiant Master 2
Ibrahim Lafi – Étudiant Master 1 Polytechnique
Alexia Le Barch – Étudiante Master 1 Polytechnique
Tharvesh Moideen Liyakat Ali – Ingénieur Bioinformatique
Charlie London – Ingénieur
Kasturi Mahadik – Post Doc
Mélanie Makhlouf – Étudiante PhD / Post Doc
Madeleine Moscatelli – Étudiante PhD / Post Doc
Damien Neuillet – Assistant ingénieur
Andrew Oldfield – Étudiant PhD
Antonio Romito – Post Doc
Olga Rosspopoff – Étudiante PhD / Post Doc
Julia Samson – Étudiante Master 2
Samantha Sheng – Post Doc
Matteo Tossolini – Étudiant Master 2
Céline Vallot – Chercheuse CNRS
Valentina Varriale – Étudiante Master 1 Polytechnique
Contacts
Claire ROUGEULLE, PhD, HDR, DR1 – CNRS
CNRS – Epigenetics and Cell Fate – UMR7216
Université Paris Cité
Bat Lamarck, 4ème étage. Case courrier 7042
35, rue Hélène BRION
75205 Paris Cedex 13
Tél : 33 (0)1 5727 8924
Fax : 33 (0)1 5727 8911
Email : claire.rougeulle@u-paris.fr
À lire aussi
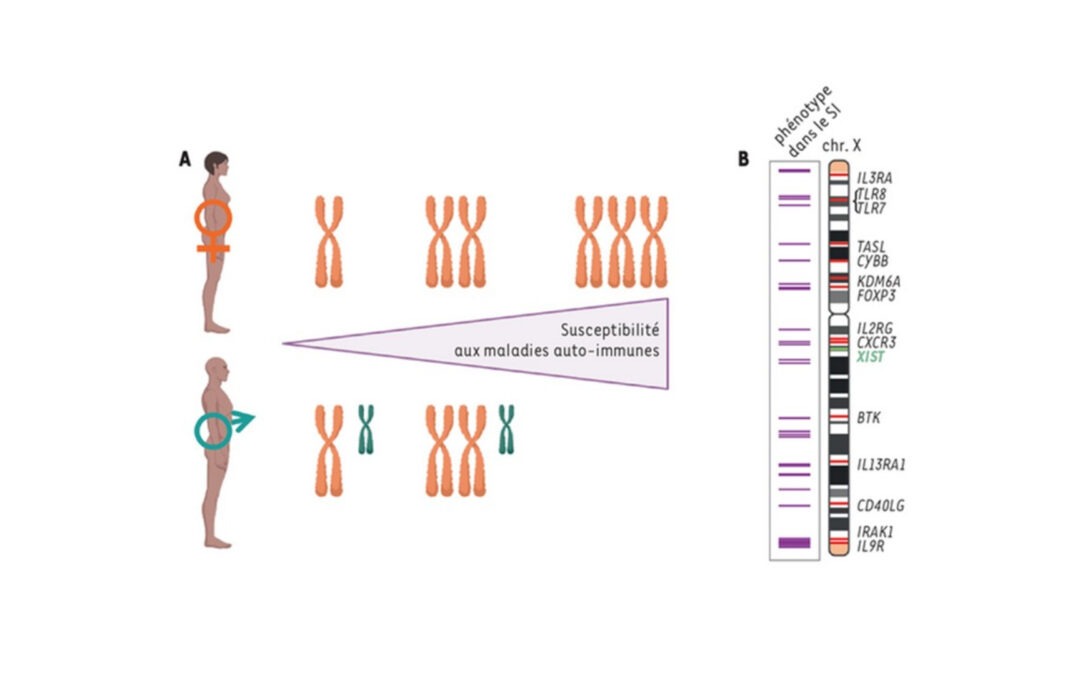
Nouvelle revue: Régulation du chromosome X et spécificités fonctionnelles des cellules femelles. Deux X valent-ils mieux qu’un ?
Et si posséder deux chromosomes X conférait aux cellules femelles des spécificités exclusives et expliquait les différences de susceptibilité des hommes et des femmes à certaines maladies ? Pourtant, l’un des chromosomes X est réprimé aléatoirement dans chaque cellule...
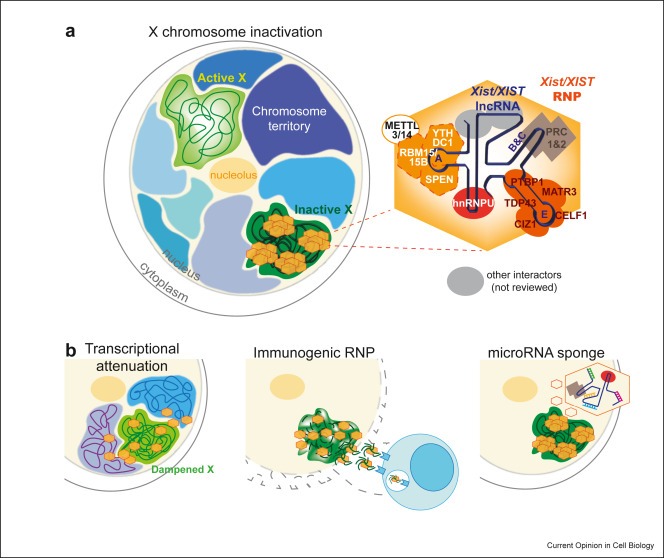
New review: Unleashing XIST from X-chromosome inactivation
The discovery that long noncoding RNAs (lncRNAs) are the most abundant gene class in the human genome has sparked significant research, particularly on Xist, a key RNA involved in X-chromosome inactivation. Recent studies have expanded our understanding of Xist's...

Félicitations Dr Carrillo
Félicitations à Léo qui a défendu son travail de thèse sur " l'exploration du lien entre l'inactivation du chromosome X et le développement des tissus extra embryonnaires ". Bravo ! © Rougeulle team À lire aussi








