Équipe Francastel
Méthylation de l’ADN et ARNs non-codants en physiopathologie

Zoom de groupe
Les 4 membres actuels de l’équipe Francastel: Claire Francastel, DR2 INSERM; Florent Hubé, CRCN CNRS; Guillaume Velasco, MCU Université Paris Cité; Katia Boyarchuk, Ingénieur CNRS.
© EDC
L’intérêt général des travaux de notre équipe concerne la multiplicité et la complexité des réseaux de régulation qui contrôlent l’expression et le maintien des génomes de mammifères, depuis le contrôle transcriptionnel ou épigénétique au niveau local jusqu’à l’organisation génomique globale dans le volume nucléaire. Les génomes des eucaryotes complexes étant transcrits de manière relativement généralisée, alors qu’ils sont constitués principalement de séquences qui ne contiennent pas d’information codante pour des protéines, une tâche importante de la biologie moderne consiste à documenter cette énorme production transcriptionnelle non codante et à comprendre sa pertinence fonctionnelle dans les réseaux de régulation qui contrôlent le destin cellulaire normal. Nos travaux visent particulièrement à déterminer si les perturbations de cette production non codante représentent une force motrice dans l’émergence de maladies et à identifier les mécanismes et facteurs responsables de son maintien dans les cellules normales. Au cours des dernières années, nous nous sommes concentrés sur des régions génomiques intrinsèquement non codantes qui représentent une large fraction des génomes de mammifères, c’est-à-dire les séquences répétées et les introns, comme paradigmes pour apporter des réponses à ces questions et comme cibles non conventionnelles des perturbations post-transcriptionnelles et épigénétiques caractéristiques de nombreuses maladies humaines.
– documenter la production transcriptionnelle non codante des régions mentionnées dans des situations normales et physiopathologiques
– caractériser la biogenèse des transcrits produits, leur maturation, leur régulation épigénétique, leur localisation sub-cellulaire et leurs complexes associés
– comprendre si et comment leur dérégulation représente une force motrice dans la maladie
– fournir une vue intégrée pan-génomique des défauts épigénétiques, transcriptionnels et d’épissage dans le contexte de défauts pathologiques de la machinerie de méthylation de l’ADN
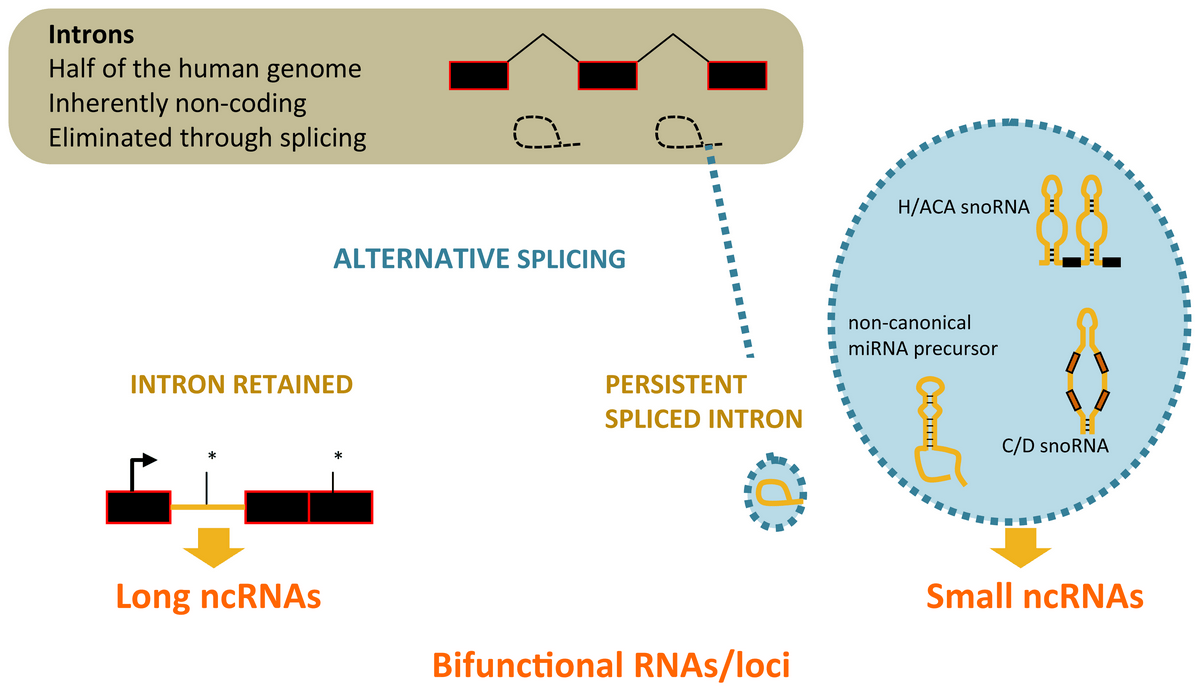
Épissage alternatif des introns et diversification de la production transcriptionnelle
Un paradigme pour tester la fonctionnalité des régions génomiques non codantes pour des protéines
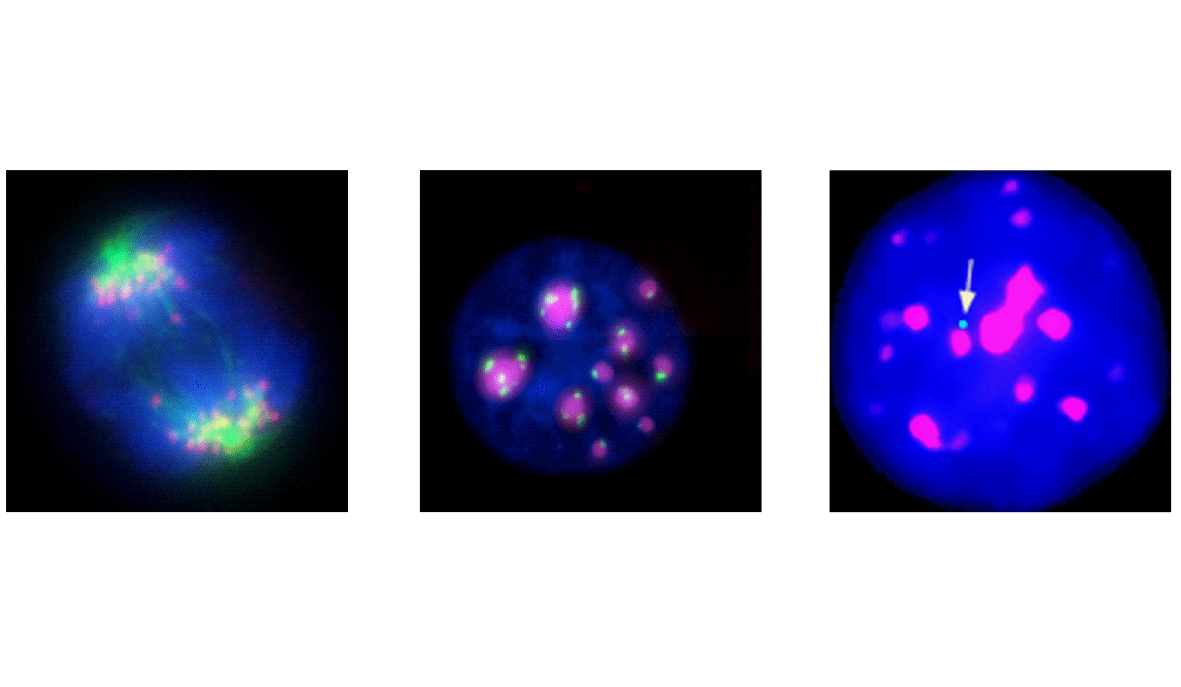
Transcription des séquences répétées centromériques
Un paradigme pour lier la transcription des séquences répétées de l’ADN à des effets moléculaires et cellulaires globaux
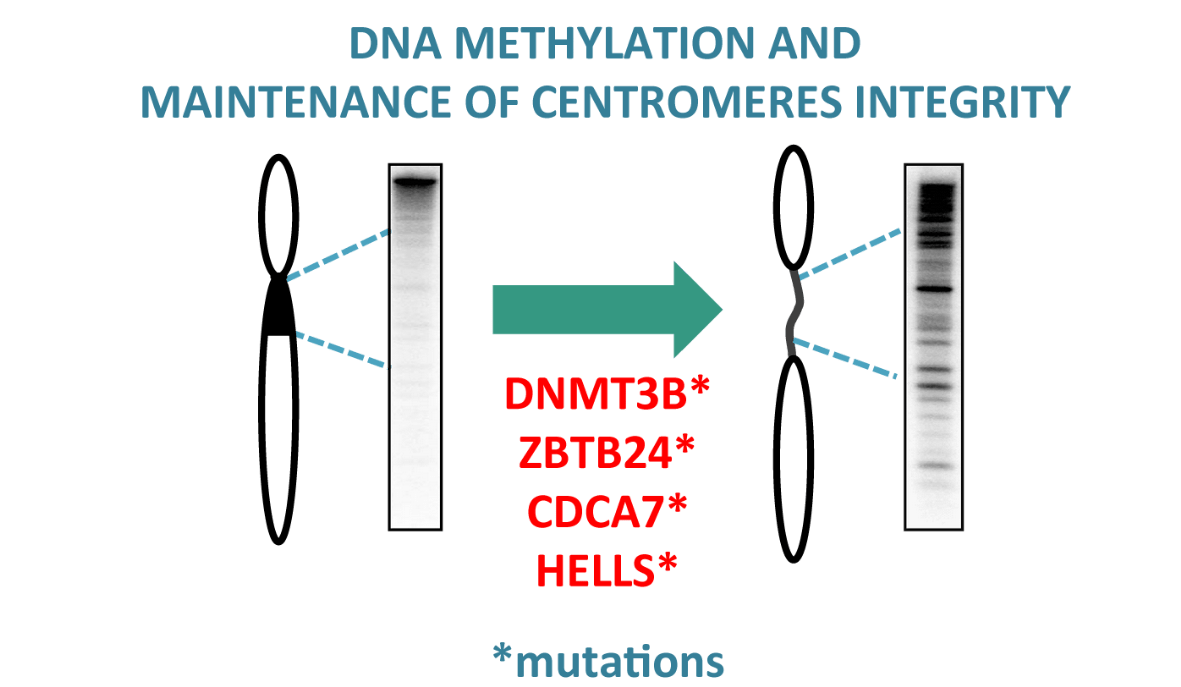
Méthylation de l’ADN et maintien de l’intégrité des génomes eucaryotes
Lorsque l’étude d’une maladie rare apporte un nouvel éclairage sur le domaine de la méthylation de l’ADN
Publications
Contact
Membres
À lire aussi

Bienvenue à Léa
Léa rejoint l’équipe en tant qu’ingénieure d’études. Après un master en virologie, elle a travaillé à Strasbourg sur les virus de la vigne, puis sur la caractérisation de la dégradation des ARNm chez les plantes à l’Institut de biologie moléculaire des plantes (IBMP)....

Sophie Polo reçoit un financement Impulscience® de la Fondation Bettencourt Schueller
Sophie Polo a reçu un financement Impulscience® pour financer un projet de recherche sur l'établissement et le maintien du chromosome X inactif en réponse aux cassures de l'ADN. C'est une formidable nouvelle pour le laboratoire! Nous remercions la Fondation...
Quand retirer une étiquette de l’ADN stoppe la croissance tumorale
Chez les mammifères, la méthylation de l’ADN est une petite modification chimique qui agit comme une étiquette sur certaines bases de l’ADN, les cytosines. Elle n’altère pas la séquence mais participe au contrôle de l’activité des gènes et la stabilité du génome. Dans...

Bienvenue à Léa, nouvelle ingénieure d’études dans l’équipe!
Léa rejoint l'équipe en tant qu'ingénieure d'études. Titulaire d'un Master en biologie moléculaire et cellulaire de Sorbonne Université, Léa va contribuer à un projet de recherche visant à disséquer les mécanismes de maintien de la méthylation de l'ADN en réponse aux...
