Méthylation de l’ADN et maintien de l’intégrité des genomes eukaryotes
Lorsque l’étude d’une maladie rare apporte un nouvel éclairage sur le domaine de la méthylation de l’ADN
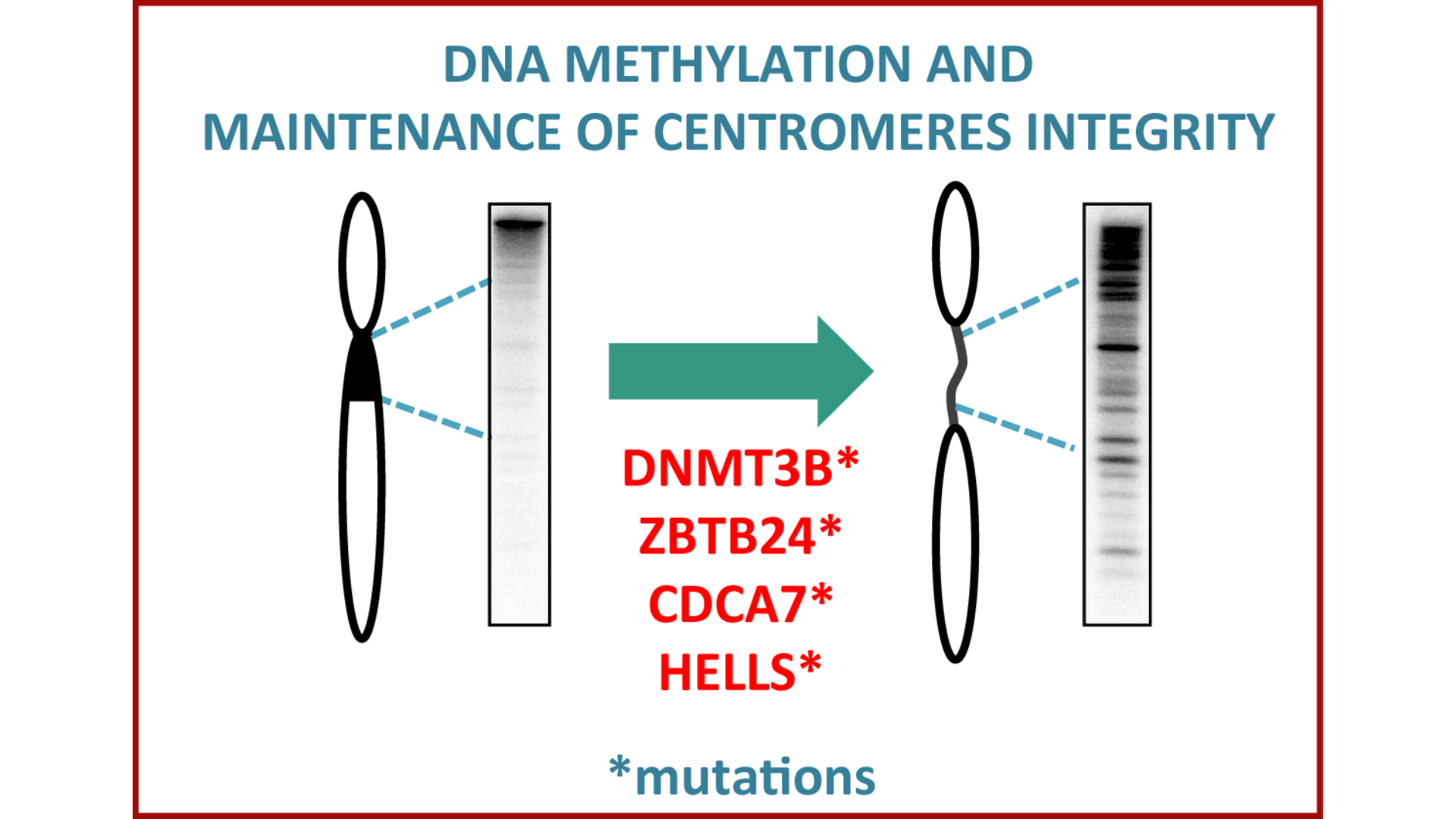
La mutation dans les 4 quatre gènes induit une hypométhylation des centromères
© EDC
La méthylation de l’ADN est parmi les modifications épigénétiques les mieux étudiées chez les vertébrés et est essentielle au développement embryonnaire normal. Compte tenu de son rôle central dans le contrôle de l’expression des gènes et de processus biologiques clés, il n’est pas surprenant que des profils de méthylation de l’ADN perturbés soient caractéristiques de nombreuses maladies humaines. Dans ce contexte, la machinerie de méthylation de l’ADN est fréquemment perturbée, bien que le lien de causalité soit parfois difficile à établir formellement. Cependant, l’existence de maladies monogéniques héréditaires qui compromettent les composants de la machinerie épigénétique offrent une occasion unique de mieux caractériser et d’aborder le lien causal avec l’émergence de perturbations moléculaires et cellulaires.
Notre intérêt pour la transcription des séquences répétées a motivé notre intérêt pour une maladie autosomique récessive rare, le syndrome ICF (Immunodéficience, instabilité Centromérique et anomalies Faciales), causée par la perte remarquable de méthylation de l’ADN au niveau des répétitions (péri) centromériques qui provoque une instabilité chromosomique. Les mutations dans l’ADN méthyltransférase DNMT3B ont été les premières causes identifiées de la maladie. L’étude de son étiologie a récemment produit avec de nouveaux candidats, dont le rôle dans la méthylation de l’ADN et le maintien de la stabilité du génome n’a jamais été suspecté avant leur implication dans la maladie.
L’implication de ces nouveaux « facteurs ICF » soulève des questions importantes concernant leur fonction, leur rôle direct ou indirect dans les voies de la méthylation de l’ADN, leurs cibles génomiques et l’impact de leurs mutations sur le transcriptome, l’épigénome et le destin cellulaire. Nous visons à fournir une vue globale et intégrée des conséquences des perturbations de la méthylation de l’ADN dans des cellules de patients et des modèles murins avec des conséquences importantes pour (i) la compréhension de la relation génotype / phénotype dans une telle maladie monogénique et épigénétique complexe, (ii) l’établissement de biomarqueurs pour faciliter le diagnostic et l’orientation de la recherche de mutations et la prise en charge appropriée les patients, et (iii) dans une perspective de recherche plus fondamentale, la compréhension des mécanismes moléculaires impliqués dans l’établissement et le maintien des patrons de méthylation de l’ADN au niveau des répétitions d’ADN ou de gènes uniques, et sur les conséquences pour la production transcriptionnelle longue ou courte, codante ou non codante, avec une pertinence additionnelle pour d’autres contextes physiopathologiques.
Publications sélectionnées :
- Gatto S, Gagliardi M, Franzese M, Leppert S, Papa M, Cammisa M, Grillo G, Velasco G, Francastel C, Toubiana S, D’Esposito M, Angelini C, Matarazzo MR. ICF-specific DNMT3B dysfunction interferes with intragenic regulation of mRNA transcription and alternative splicing . Nucleic Acids Res. 2017 Mar 9; 45(10):5739-5756. PMID: 28334849
- Sagie S, Toubiana S, Hartono S, Katzir H, Tzur-Gilat A, Havazelet S, Francastel C, Velasco G, Chedin F, Selig S. Telomeres in ICF syndrome cells are vulnerable to DNA damage due to elevated DNA:RNA hybrids. Nat Commun. 2017 Jan 24;8:14015. PMID: 28117327
- Sterlin D, Velasco G, Moshous D, Touzot F, Mahlaoui N, Fischer A, Suarez F, Francastel C, Picard C. Genetic, Cellular and Clinical Features of ICF Syndrome: a French National Survey . J Clin Immunol. 2016 Feb;36(2):149-59. PMID: 26851945
- Thijssen PE, Ito Y, Grillo G, Wang J, Velasco G, Nitta H, Unoki M, Yoshihara M, Suyama M, Sun Y, Lemmers RJ, de Greef JC, Gennery A, Picco P, Kloeckener-Gruissem B, Güngör T, Reisli I, Picard C, Kebaili K, Roquelaure B, Iwai T, Kondo I, Kubota T, van Ostaijen-Ten Dam MM, van Tol MJ, Weemaes C, Francastel C, van der Maarel SM, Sasaki H. Mutations in CDCA7 and HELLS cause immunodeficiency-centromeric instability-facial anomalies syndrome. Nat Commun. 2015 Jul 28;6:7870. PMID: 26216346
- Walton E, Francastel C, Velasco G. Dnmt3b prefers germ line genes and centromeric regions: lessons from ICF and cancer and implications for diseases. Biology (Basel). 2014 Sep 5;3(3):578-605. PMID: 25198254
- Velasco G, Walton EL, Sterlin D, Hédouin S, Nitta H, Yuya I, Fouyssac F, Mégarbané A, Sasaki H, Picard C, Francastel C. Germline genes hypomethylation and expression define a molecular signature in peripheral blood of ICF patients: implications for diagnosis and etiology. Orphanet J Rare Dis. 2014 Apr 17;9(1):56. PMID: 24742017
- Nitta H, Unoki M, Ichiyanagi K, Kosho T, Shigemura T, Takahashi H, Velasco G, Francastel C, Picard C, Kubota T, Sasaki H. Three novel ZBTB24 mutations identified in Japanese and Cape Verdean type 2 ICF syndrome patients. J Hum Genet. 2013 Jul;58(7):455-60. PMID: 23739126
- Walton EL, Francastel C, Velasco G. Maintenance of DNA methylation: Dnmt3b joins the dance. Epigenetics. 2011 Nov;6(11):1373-7. PMID: 22048250
- Velasco G, Hubé F, Rollin J, Neuillet D, Philippe C, Bouzinba-Segard H, Galvani A, Viegas-Péquignot E, Francastel C. Dnmt3b recruitment through E2F6 transcriptional repressor mediates germ-line gene silencing in murine somatic tissues. Proc Natl Acad Sci U S A. 2010 May 18;107(20):9281-6. PMID: 20439742
À lire aussi
Theileria annulata et Cancer : une stratégie du parasite dévoilée !

Séminaire Laetitia Aubry – 24 avril 2024
Mercredi 24 avril 2024, à 11h. Laetitia Aubry Université d’Evry / I-StemInvitée par Valérie Drouet “Harnessing pluripotent stem cells derivatives from patients with Wolfram syndrome to reveal pathological mechanisms and identify potential therapeutic chemical...

Séminaire Moussa Benhamed – 17 avril 2024
Mercredi 17 avril 2024, à 11h. Moussa Benhamed Institut des Sciences des Plantes, Paris SaclayInvité par Valérie Mezger “Exploring the chromatin-based regulation of enhancer-promoter contact and its impact on gene expression” Le séminaire se tiendra à l’Institut...

Afterwork professionnel – 22 avril 2024
Savez-vous ce qui existe en dehors du monde académique ?Venez en parler avec nos deux invités le lundi 22 avril 2024 à 18h15. Où ? Unité Épigénétique et Destin Cellulaire (EDC)Université Paris Cité - 35 Rue Hélène Brion 75013 Paris - Bâtiment Lamarck B - Hall...
