EpiG : Epigénomique fonctionnelle
La plateforme Epigénomique Fonctionnelle a pour objectifs d’assurer la mise à disposition d’équipements et de former le personnel à leur utilisation.
Elle propose des prestations de services permettant l’analyse de l’épigénome et participe aux développement de nouvelles techniques en fonction des évolutions du domaine.
La plateforme Epigénomique Fonctionnelle est ouverte à la communauté scientifique académique et industrielle.
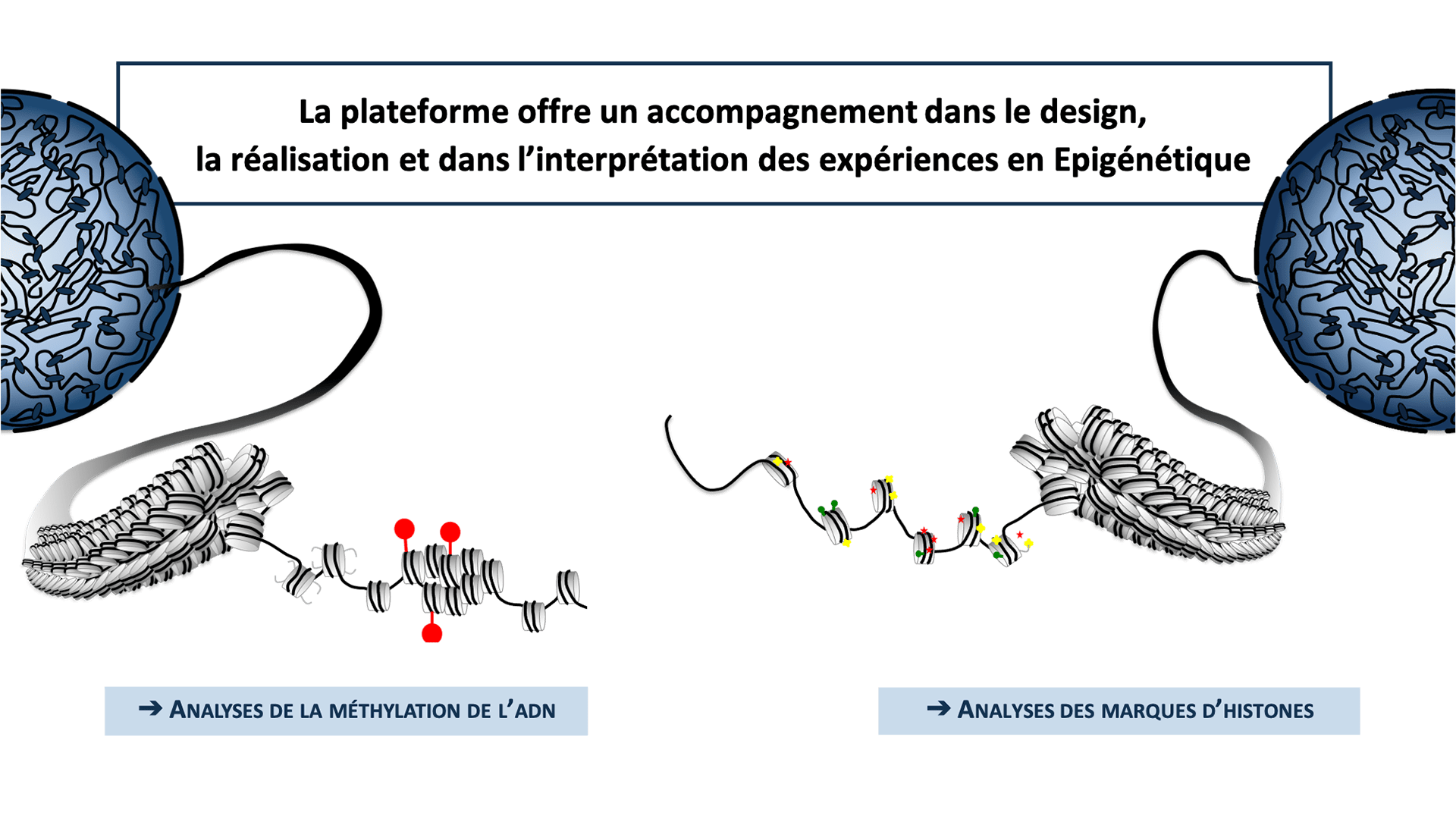
Schéma des expertises de la plateforme
La plateforme offre un accompagnement dans le design, la réalisation et dans l’interprétation des expériences en Epigénétique.
© Laure Ferry
Expertises
La plateforme Epigénomique Fonctionnelle offre une expertise complète pour l’analyse des marques épigénétiques. Elle évalue la faisabilité des projets déposés et propose la méthode la plus adaptée pour leur aboutissement.
Méthylation de l’ADN
- LUMA (LUminometric Methylation Assay)
- MeDIP ou hMeDIP (Methylated ou hydroxyMethylated Immunoprecipitation Assay)
- Bisulfite pyroséquençage
Marques post-tranductionnelles des Histones
- Cut & Run
- ChIP
Soumettre un projet
Vous pouvez nous contacter pour que nous puissions répondre à vos questions et discuter ensemble de votre projet.
Vous devez remplir la fiche de demande de projet et l’envoyer à l’adresse suivante : parisepigenetics.pf@gmail.com
Développements
La plateforme Epigénomique Fonctionnelle s’implique dans le développement de nouvelles techniques d’analyse de l’Epigénome :
- Automatisation Cut & Run
- Cut & Tag
- Enzymatic Methyl-seq ( EM-seq)
- Nanopore
Analyses épigénétiques proposées
Les prestations de service disponibles pour l’analyse de la méthylation de l’ADN et des marques d’histones.
Équipements
Les équipements disponibles sur la plateforme Epigénomique Fonctionnelle.
Membres
Responsable scientifique : Pierre-Antoine Defossez
Responsable technique : Laure Ferry
Comité de Pilotage :
Publications
- Planques A, Pierre K, Ferry L, Grunau C, Gazave E, Vervoort M. DNA methylation atlas and machinery in the developing and regenerating annelid Platynereis dumerilii. BMC Biology, Vol 19, Iss 1, Pp 1-26, 2021. DOI: 10.1186/s12915-021-01074-5
- Marchal C, de Dieuleveult M, Saint-Ruf C, Guinot N, Ferry L, Olalla Saad ST, Lazarini M, Defossez PA, Miotto B. Depletion of ZBTB38 potentiates the effects of DNA demethylating agents in cancer cells via CDKN1C mRNA up-regulation. Oncogenesis; Oct 2018, Vol. 7 Issue 10, p1-18, 18p DOI: 10.1038/s41389-018-0092-0
- Velasco G, Grillo G, Touleimat N, Ferry L, Ivkovic I, Ribierre F, Deleuze JF, Chantalat S, Picard C, Francastel C. Comparative methylome analysis of ICF patients identifies heterochromatin loci that require ZBTB24, CDCA7 and HELLS for their methylated state. Human Molecular Genetics, 2018 Jul 15;27(14):2409-2424. DOI: 10.1093/hmg/ddy130.
- Ferry L, Fournier A, Tsusaka T, Adelmant G, Shimazu T, Matano S, Kirsh O, Amouroux R, Dohmae N, Suzuki T, Filion GJ, Deng W, de Dieuleveult M, Fritsch L, Kudithipudi S, Jeltsch A, Leonhardt H, Hajkova P, Marto JA, Arita K, Shinkai Y, Defossez PA. Methylation of DNA Ligase 1 by G9a/GLP Recruits UHRF1 to Replicating DNA and Regulates DNA Methylation. Molecular Cell. August 17, 2017, Vol. 67 Issue 4, p550. DOI: 10.1016/j.molcel.2017.07.012
- Kebir O, Chaumette B, Rivollier F, Miozzo F, Lemieux Perreault LP, Barhdadi A, Provost S, Plaze M, Bourgin J, the ICAAR team, Gaillard R, Mezger V, Dubé M-P , Krebs M-O. Methylomic changes during conversion to psychosis. Molecular Psychiatry (2017) 22, 512-518. DOI: 10.1038/mp.2016.53.
Contact
Vous pouvez nous contacter par mail à l’adresse suivante : parisepigenetics.pf@gmail.com
Plateforme Epigénomique Fonctionnelle
35 rue Hélène Brion
Bâtiment Lamarck B, RB77-81
75013 PARIS
+33(0)1 57 27 89 26
À lire aussi

Bienvenue à Léa
Léa rejoint l’équipe en tant qu’ingénieure d’études. Après un master en virologie, elle a travaillé à Strasbourg sur les virus de la vigne, puis sur la caractérisation de la dégradation des ARNm chez les plantes à l’Institut de biologie moléculaire des plantes (IBMP)....

Sophie Polo reçoit un financement Impulscience® de la Fondation Bettencourt Schueller
Sophie Polo a reçu un financement Impulscience® pour financer un projet de recherche sur l'établissement et le maintien du chromosome X inactif en réponse aux cassures de l'ADN. C'est une formidable nouvelle pour le laboratoire! Nous remercions la Fondation...
Quand retirer une étiquette de l’ADN stoppe la croissance tumorale
Chez les mammifères, la méthylation de l’ADN est une petite modification chimique qui agit comme une étiquette sur certaines bases de l’ADN, les cytosines. Elle n’altère pas la séquence mais participe au contrôle de l’activité des gènes et la stabilité du génome. Dans...

Bienvenue à Léa, nouvelle ingénieure d’études dans l’équipe!
Léa rejoint l'équipe en tant qu'ingénieure d'études. Titulaire d'un Master en biologie moléculaire et cellulaire de Sorbonne Université, Léa va contribuer à un projet de recherche visant à disséquer les mécanismes de maintien de la méthylation de l'ADN en réponse aux...
