Méthylation de l’ADN dans les cellules souches
L’un de nos axes de recherche vise à comprendre comment les schémas de méthylation de l’ADN sont établis, maintenus et interprétés au cours du développement embryonnaire. Pour répondre à ces questions, nous utilisons des cellules souches embryonnaires (ES) de souris comme modèle, et nous appliquons un large éventail d’outils : biologie cellulaire, génomique, protéomique, bioinformatique… Cela nous a par exemple permis de déchiffrer d’importants mécanismes de mémoire épigénétique (Ferry et al, Mol Cell 2017, review in Petryk et al NAR 2020). Plus récemment, nous avons réalisé des cribles CRISPR à l’échelle du génome pour identifier les facteurs reliant l’épigénétique et l’état cellulaire (Gupta, Yakhou et al., NSMB 2023).
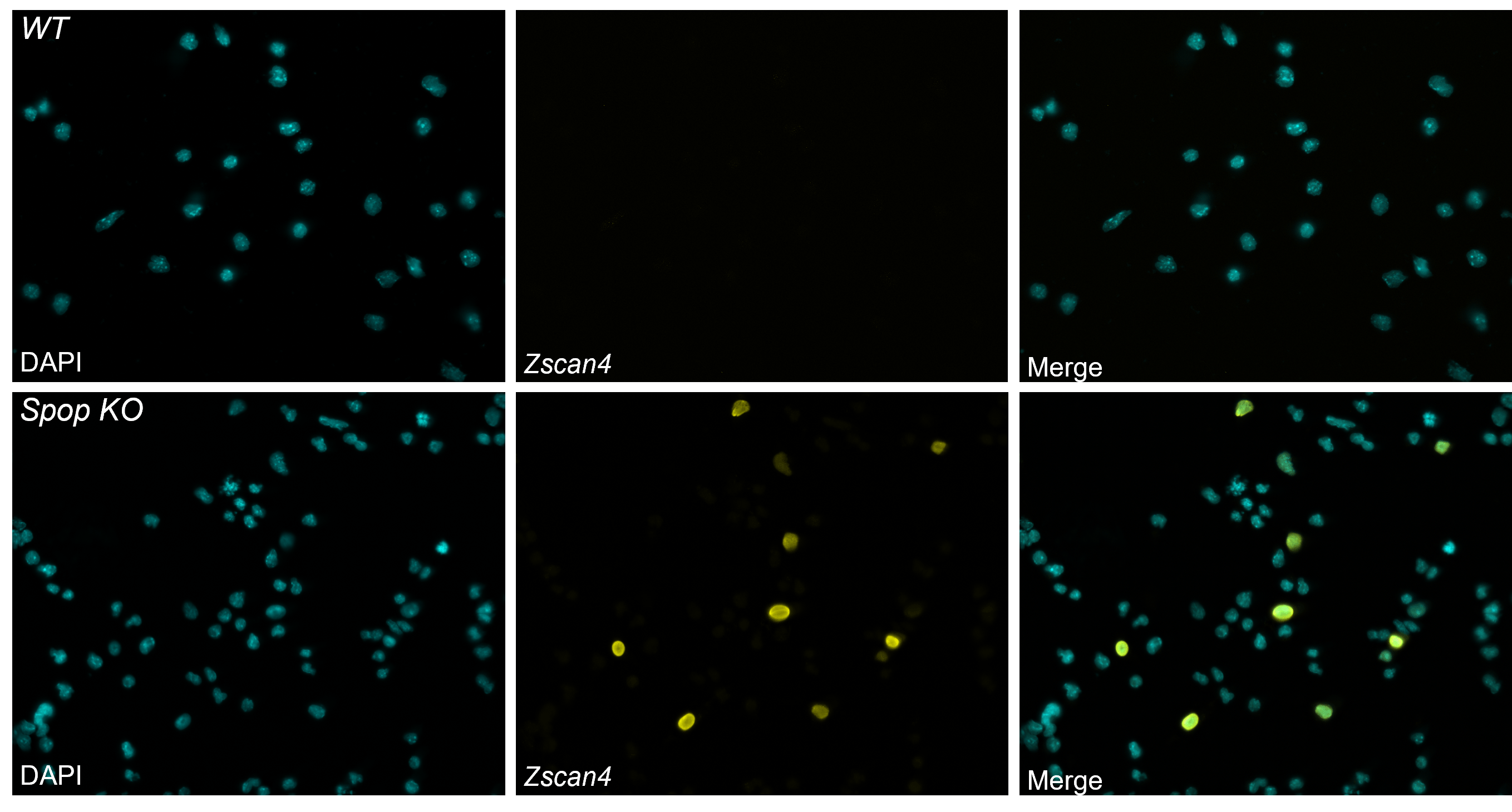
Un crible CRISPR a permis d’identifier de nouveaux répresseurs de l’état totipotent dans les cellules souches embryonnaires murines
Chez les mammifères, seuls le zygote et les blastomères de l’embryon précoce sont totalement totipotents. Cette totipotence est reflétée in vitro par l’expression de marqueurs tels que Zscan4. Nous avons réalisé un crible CRISPR KO à l’échelle du génome dans des cellules souches embryonnaires murines, à la recherche de mutants qui réactivent l’expression des marqueurs de totipotence, comme par exemple le knock-out du gène Spop.
À lire aussi

Félicitations à Delphine et Julia pour le financement de leurs projets de recherche
Félicitations à Delphine Burlet pour l'obtention d'un financement postdoctoral de la Fondation de France et à Julia Roche Dupuy pour avoir décroché une bourse de l'école doctorale Hématologie-Oncogénèse-Biothérapies pour financer sa thèse. Delphine Burlet, Julia Roche...

Bienvenue à Annabelle, nouvelle post-doctorante dans l’équipe!
Annabelle rejoint le laboratoire en tant que post-doctorante. Elle a réalisé sa thèse à l'Université d'Oxford sous la direction de Monika Gullerova. Ses travaux de doctorat ont porté sur le rôle des petits ARNs non codants de type Y et de la protéine qui les lie,...

Bienvenue à Orlane
Aujourd'hui, nous accueillons un nouveau membre dans l'équipe : Orlane. Elle est étudiante en première année du Brevet de Technicien Supérieur Biotechnologies en Recherche et Production.Orlane travaillera avec Jérémy sur la fonction et la caractérisation des protéines...

Financements de 4e année de thèse pour Anaëlle
Félicitations à Anaëlle Azogui pour l’obtention de financements de 4e année de thèse par la fondation ARC (fondation pour la recherche sur le cancer). À lire aussi
