Épissage alternatif des introns et diversification de la production transcriptionnelle
Un paradigme pour tester la fonctionnalité des régions génomiques non codantes pour des protéines
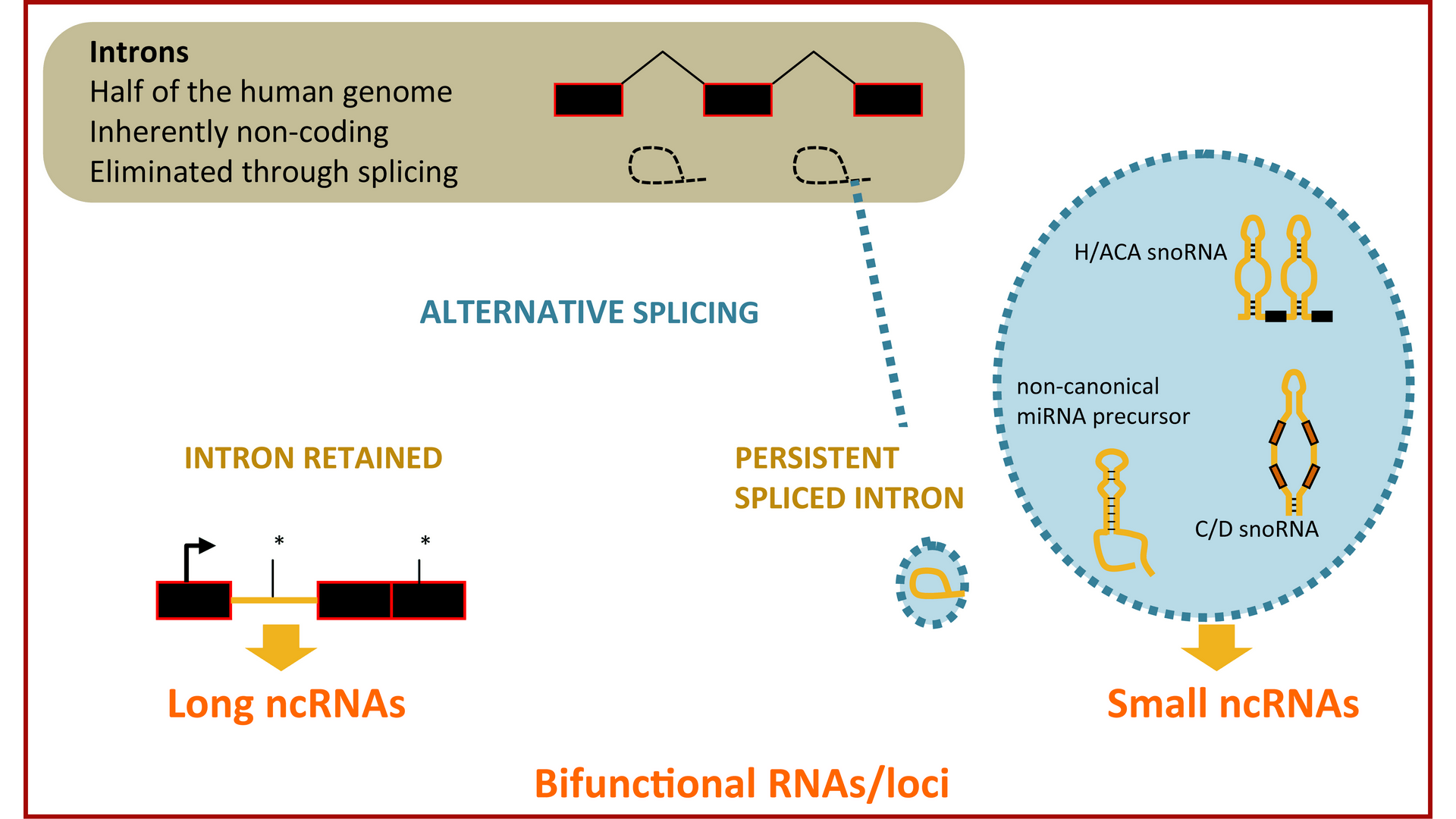
ARN bifonctionnel
© EDC
Les introns représentent près de la moitié du génome humain bien que, pour leur grande majorité, ils soient éliminés des transcrits eucaryotes par épissage de l’ARN. Pourtant, des informations importantes sont intégrées dans les introns, la plus remarquable étant la libération de différentes classes de petits ARNnc régulateurs directement à partir de l’épissage. En outre, nous avons montré que leur excision ou leur rétention, selon le contexte cellulaire, contribue à la diversification de l’information portée par les gènes en produisant des ARN fonctionnels au lieu d’un ARNm codant pour des protéines.
Une conséquence importante est que l’épissage alternatif fonctionne comme un commutateur développemental flexible qui confère une certaine plasticité à la production transcriptionnelle des génomes eucaryotes, permettant de diversifier non seulement le protéome mais aussi le transcriptome.
Nous explorons l’épissage alternatif des introns comme mécanisme permettant d’affiner la production d’ARNm, de longs ARNnc ou de petits ARNnc régulateurs dérivés d’intron (que nous avons appelé SID pour Short Intron-Derived) au cours de la différenciation musculaire normale et pathologique où l’épissage est altéré, comme c’est le cas dans la Dystrophie Myotonique de type 1 (DM1). Nous utilisons des prédictions bioinformatiques et le séquençage d’ARN à haut débit et validons expérimentalement les ARNnc différentiels dans des systèmes modèles.
Publications sélectionnées:
- Bogard B*, Tellier G*, Francastel C, Hubé F. A Tool to Design Bridging Oligos Used to Detect Pseudouridylation Sites on RNA after CMC Treatment. Non-coding RNA, 2022, 8(5):63. PMID: 36287115
- Bogard B, Francastel C, Hubé F. Multiple information carried by RNAs: total eclipse or a light at the end of the tunnel? RNA Biol, 2020, 17(12):1707-1720. PMID: 32559119
- Hubé F, Francastel C. Coding and non-coding RNAs, the frontier has never been so blurred. Frontiers in Genetics, 2018, 9:140. PMID: 29720998
- Bogard B, Francastel C, Hubé F. A new method for the identification of thousands of circular RNAs. Non-coding RNA Investig, 2018, 2:5. Site: Fulltext
- Hubé F, Ulveling D, Sureau A, Forveille S, Francastel C. Short intron-derived ncRNAs. Nucleic Acids Res. 2017 Jan 3;45(8):4768-4781. PMID: 28053119
- Hubé F, Francastel C. “Pocket-sized RNA-Seq”: a method to capture new mature microRNA produced from a genomic region of interest. Non-coding RNA. 2015 June;1(2):127-138. Paper here
- Hubé F, Francastel C. Mammalian Introns: When the Junk Generates Molecular Diversity. Int J of Mol Sci. 2015 Feb 20;16(3):4429-4452. PMID: 25710723
- Ulveling D, Dinger M, Francastel C, Hubé F. Identification of a dinucleotide signature that discriminates coding from non-coding long RNAs. Front Genet. 2014 Sep 9;5:316. PMID: 25250049
- Ulveling D, Francastel C, Hubé F. When one is better than two: RNA with dual functions. Biochimie. 2011 Apr;93(4):633-44. Review. PMID: 21111023
- Ulveling D, Francastel C, Hubé F. Identification of potentially new bifunctional RNA based on genome-wide data-mining of alternative splicing events. Biochimie. 2011 Nov;93(11):2024-7. PMID: 21729736
- Hubé F, Velasco G, Rollin J, Furling D, Francastel C. Steroid receptor RNA activator protein binds to and counteracts SRA RNA-mediated activation of MyoD and muscle differentiation. Nucleic Acids Res. 2011 Jan;39(2):513-25. PMID: 20855289
À lire aussi

Bienvenue à Léa
Léa rejoint l’équipe en tant qu’ingénieure d’études. Après un master en virologie, elle a travaillé à Strasbourg sur les virus de la vigne, puis sur la caractérisation de la dégradation des ARNm chez les plantes à l’Institut de biologie moléculaire des plantes (IBMP)....

Sophie Polo reçoit un financement Impulscience® de la Fondation Bettencourt Schueller
Sophie Polo a reçu un financement Impulscience® pour financer un projet de recherche sur l'établissement et le maintien du chromosome X inactif en réponse aux cassures de l'ADN. C'est une formidable nouvelle pour le laboratoire! Nous remercions la Fondation...
Quand retirer une étiquette de l’ADN stoppe la croissance tumorale
Chez les mammifères, la méthylation de l’ADN est une petite modification chimique qui agit comme une étiquette sur certaines bases de l’ADN, les cytosines. Elle n’altère pas la séquence mais participe au contrôle de l’activité des gènes et la stabilité du génome. Dans...

Bienvenue à Léa, nouvelle ingénieure d’études dans l’équipe!
Léa rejoint l'équipe en tant qu'ingénieure d'études. Titulaire d'un Master en biologie moléculaire et cellulaire de Sorbonne Université, Léa va contribuer à un projet de recherche visant à disséquer les mécanismes de maintien de la méthylation de l'ADN en réponse aux...
