Analyses épigénétiques proposées
Selon la question d’intérêt, la plateforme Epigénomique propose différentes techniques pour l’analyse de la méthylation de l’ADN et des marques d’histones.
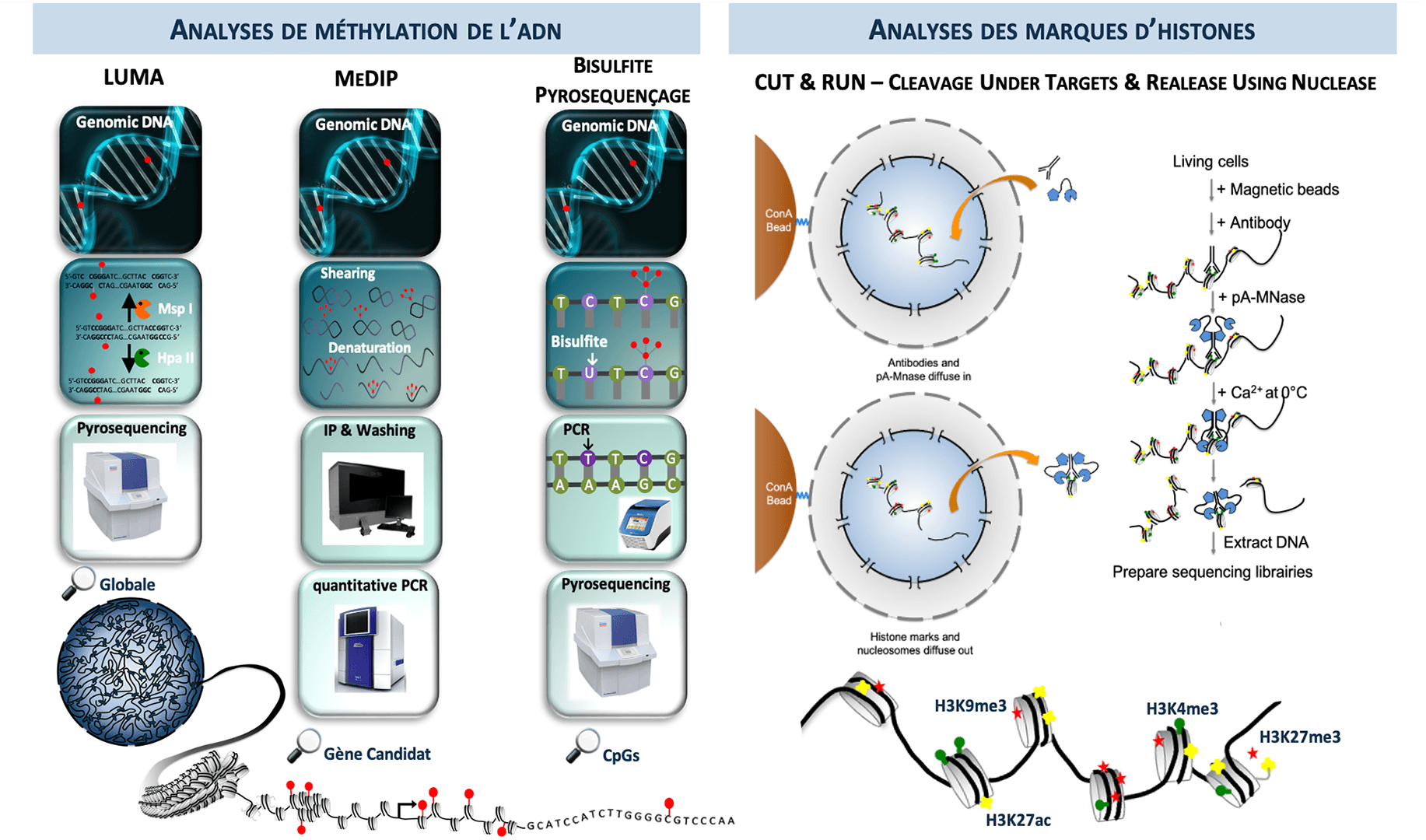
Schéma des techniques proposées sur la plateforme Epigénomique.
La plateforme Epigénomique propose des techniques pour l’analyse de la méthylation de l’ADN et des marques d’histones
© Laure Ferry
Analyses de méthylation de l’ADN
LUMA (LUminometric Methylation Assay)
-
Méthode pour l’analyse globale de la méthylation de l’ADN
- Quantification du nombre de coupure par des enzymes de restriction, sensibles ou non à la méthylation
- Détermination du niveau de méthylation : rapide et quantitative.
- Aucune information concernant la localisation ou la répartition de la méthylation de l’ADN.
MeDIP (Methylated DNA ImmunoPrécipitation)
- Enrichissement d’ADN génomique fragmenté en utilisant un anticorps spécifique de la 5mC.
-
Les régions présenatnt des faibles densités de CpG ne peuvent pas être analysées précisément par MeDIP.
-
L’anticorps 5mC permet de discriminer les modifications 5-hmC et 5-mC.
Bisulfite Pyroséquençage
- Analyse et quantification du niveau de méthylation d’un ou plusieurs CpG consécutifs.
-
Méthode basée sur la conversion bisulfite qui ne permet pas de différencier 5-mC et 5-hmC.
Analyses des marques d’histones
CUT & RUN (Cleavage Under Targets & Release Using Nuclease)
- Technique utilisée pour cibler les interactions protéine-ADN dans le contexte chromatinien naturel de la cellule.
- Méthode rapide et robuste avec peu de cellules faibles.
- Ne nécessite pas de fixation au formaldéhyde, de fragmentation de la chromatine et d’immunoprécipitation, ce qui en fait une méthode plus efficace pour identifier les gènes cibles.
À lire aussi

Bienvenue à Léa
Léa rejoint l’équipe en tant qu’ingénieure d’études. Après un master en virologie, elle a travaillé à Strasbourg sur les virus de la vigne, puis sur la caractérisation de la dégradation des ARNm chez les plantes à l’Institut de biologie moléculaire des plantes (IBMP)....

Sophie Polo reçoit un financement Impulscience® de la Fondation Bettencourt Schueller
Sophie Polo a reçu un financement Impulscience® pour financer un projet de recherche sur l'établissement et le maintien du chromosome X inactif en réponse aux cassures de l'ADN. C'est une formidable nouvelle pour le laboratoire! Nous remercions la Fondation...
Quand retirer une étiquette de l’ADN stoppe la croissance tumorale
Chez les mammifères, la méthylation de l’ADN est une petite modification chimique qui agit comme une étiquette sur certaines bases de l’ADN, les cytosines. Elle n’altère pas la séquence mais participe au contrôle de l’activité des gènes et la stabilité du génome. Dans...

Bienvenue à Léa, nouvelle ingénieure d’études dans l’équipe!
Léa rejoint l'équipe en tant qu'ingénieure d'études. Titulaire d'un Master en biologie moléculaire et cellulaire de Sorbonne Université, Léa va contribuer à un projet de recherche visant à disséquer les mécanismes de maintien de la méthylation de l'ADN en réponse aux...
