Équipe Defossez
Dynamique et interprétation de la méthylation de l’ADN chez les mammifères

Equipe DEFOSSEZ
L’équipe du Dr Pierre-Antoine Defossez s’intéresse aux mécanismes épigénétiques de régulation de l’expression des gènes chez les mammifères.
Épigénétique : importance en biologie et en santé humaine
L’épigénétique est l’étude des changements de l’expression des gènes qui n’impliquent pas de modifications de la séquence d’ADN correspondante. Ces modifications peuvent inclure la fixation de groupes chimiques (tels que les groupes méthyles) à la molécule d’ADN ou aux protéines histones autour desquelles l’ADN est enroulé pour former les chromosomes. Ces modifications peuvent “activer” ou “désactiver” les gènes et affecter la façon dont les cellules lisent les instructions des gènes.
L’épigénétique est importante car elle permet d’expliquer comment l’environnement peut affecter la façon dont les gènes sont exprimés, et comment ces changements peuvent être transmis d’une génération à l’autre. Par exemple, des études ont montré que certains facteurs environnementaux, tels que l’alimentation et le stress, peuvent entraîner des modifications des marques épigénétiques sur les gènes d’un individu. Ces changements peuvent par la suite affecter la santé de l’individu et augmenter son risque de développer certaines maladies.
L’épigénétique est également importante dans le domaine de la médecine car elle pourrait fournir de nouvelles cibles thérapeutiques. Par exemple, les médicaments qui ciblent les enzymes impliquées dans l’établissement ou la suppression des marques épigénétiques pourraient être utilisés pour traiter le cancer ou d’autres maladies causées par des changements dans l’expression des gènes. En outre, la compréhension de l’épigénétique pourrait aider les chercheurs à comprendre comment prévenir certaines maladies en identifiant les facteurs environnementaux qui affectent les marques épigénétiques d’un individu.
La méthylation de l’ADN : une marque épigénétique paradigmatique aux fonctions clés
La méthylation de l’ADN est un processus par lequel des groupes méthyles sont ajoutés à la molécule d’ADN. Ce processus joue un rôle important dans les cellules souches, car il contribue à réguler l’équilibre entre l’autorenouvellement et la différenciation. Dans les cellules souches, la méthylation de l’ADN contribue à inhiber l’expression les gènes qui favorisent la différenciation, ce qui permet aux cellules souches de conserver leur capacité à se diviser et à produire davantage de cellules souches. De plus, la méthylation de l’ADN est également importante dans le processus de reprogrammation des cellules adultes en cellules souches pluripotentes induites, qui ont la capacité de se différencier en n’importe quel type de cellule dans le corps. Dans le cas du cancer, la méthylation de l’ADN est souvent associée à la répression des gènes suppresseurs de tumeurs, qui sont responsables de la prévention de la croissance incontrôlée des cellules. Lorsque ces gènes sont réprimés par la méthylation de l’ADN, les cellules cancéreuses peuvent continuer à se diviser et à se développer sans aucune restriction. De plus, les cellules cancéreuses présentent également une hypométhylation globale de leur génome, ce qui signifie que l’ADN des cellules cancéreuses est moins méthylé que celui des cellules normales. Cette hypométhylation globale peut contribuer au développement de cancer en perturbant la régulation de nombreux gènes impliqués dans la croissance et la division cellulaires. Des médicaments appelés inhibiteurs de l’ADN méthyltransférase sont mis au point pour cibler ce processus et sont utilisés comme traitement du cancer.
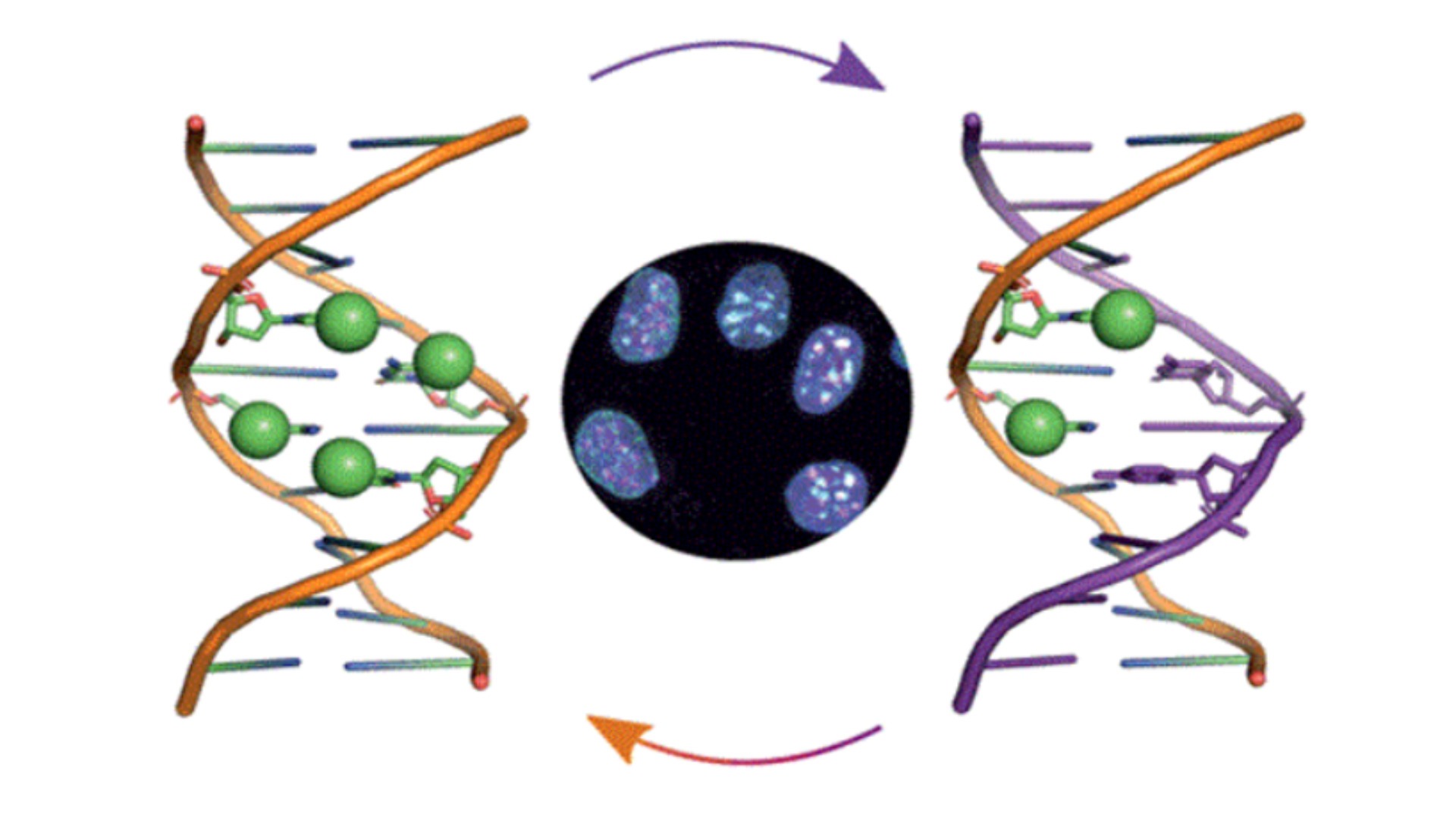
Méthylation de l’ADN dans les cellules souches
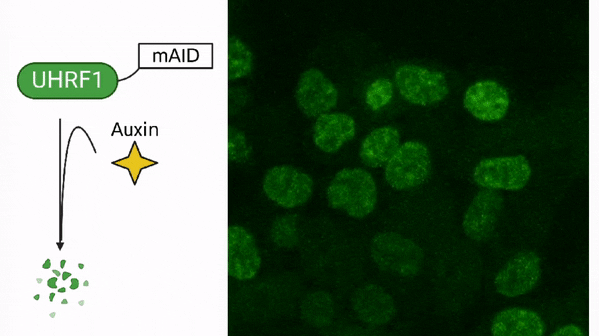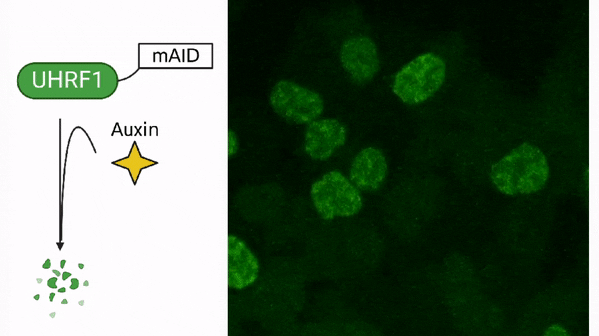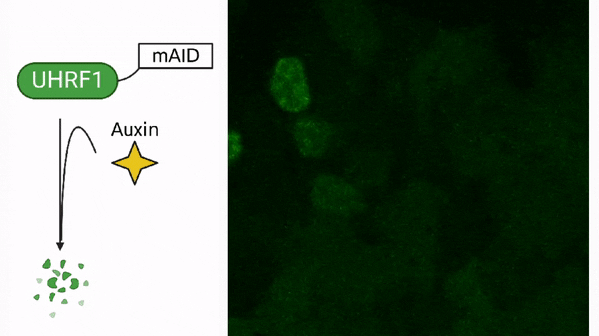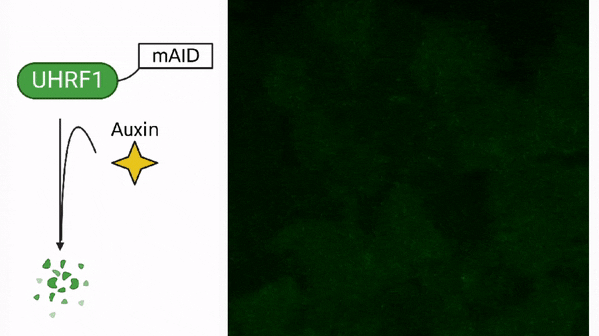
Méthylation de l’ADN dans le cancer
Publications
Yamaguchi K, Chen X, Rodgers B, et al. Non-canonical functions of UHRF1 maintain DNA methylation homeostasis in cancer cells. Nat Commun 15, 2960 (2024). doi:10.1038/s41467-024-47314-4
Marthe Laisné, Sarah Benlamara, André Nicolas, et al. Cancer/Testis genes are predictive of breast tumor subtypes. Oncogene. 2024 May;43(18):1369-1385. doi: 10.1038/s41388-024-03002-7
Lanciano S, Philippe C, Sarkar A, et al. Comprehensive locus-specific L1 DNA methylation profiling reveals the epigenetic and transcriptional interplay between L1s and their integration sites. Cell Genomics. 2024 Jan 30:100498. doi: 10.1016/j.xgen.2024.100498
Chua BH, Zaal Anuar N, Ferry L, et al. E4F1 and ZNF148 are transcriptional activators of the -57A > C and wild-type TERT promoter. Genome Res. Published online November 2, 2023. doi:10.1101/gr.277724.123
Yakhou L, Azogui A, Gupta N, et al. A genetic screen identifies BEND3 as a regulator of bivalent gene expression and global DNA methylation. Nucleic Acids Res. Published online August 31, 2023:gkad719. doi:10.1093/nar/gkad719
Gupta N, Yakhou L, Albert JR, et al. A genome-wide screen reveals new regulators of the 2-cell-like cell state. Nat Struct Mol Biol. 2023;30(8):1105-1118. doi:10.1038/s41594-023-01038-z
Kikuchi, Amika, Hiroki Onoda, Kosuke Yamaguchi, Satomi Kori, Shun Matsuzawa, Yoshie Chiba, Shota Tanimoto, et al. “Structural Basis for Activation of DNMT1.” Nature Communications 13, no. 1 (November 21, 2022): 7130, doi: 10.1038/s41467-022-34779-4
Marchal, P.-A. Defossez, and B. Miotto, ‘Context-dependent CpG methylation directs cell-specific binding of transcription factor ZBTB38’, Epigenetics, pp. 1–22, Aug. 2022, doi: 10.1080/15592294.2022.2111135.
Yamaguchi, X. Chen, A. Oji, I. Hiratani, and P.-A. Defossez, ‘Large-Scale Chromatin Rearrangements in Cancer’, Cancers (Basel), vol. 14, no. 10, p. 2384, May 2022, doi: 10.3390/cancers14102384.
Kori S, Shibahashi Y, Ekimoto T, Nishiyama A, Yoshimi S, Yamaguchi K, Nagatoishi S, Ohta M, Tsumoto K, Nakanishi M, Defossez PA, Ikeguchi M, Arita K. Structure-based screening combined with computational and biochemical analyses identified the inhibitor targeting the binding of DNA Ligase 1 to UHRF1. Bioorg Med Chem. 2021 Dec 15;52:116500. doi: 10.1016/j.bmc.2021.116500. Epub 2021 Nov 10.
Petryk N, Reverón-Gómez N, González-Aguilera C, Dalby M, Andersson R, Groth A. Genome-wide and sister chromatid-resolved profiling of protein occupancy in replicated chromatin with ChOR-seq and SCAR-seq. Nat Protoc. 2021 Sep;16(9):4446-4493. doi: 10.1038/s41596-021-00585-3.
Blin M, Lacroix L, Petryk N, Jaszczyszyn Y, Chen CL, Hyrien O, Le Tallec B. DNA molecular combing-based replication fork directionality profiling. Nucleic Acids Res. 2021 Jul 9;49(12):e69. doi: 10.1093/nar/gkab219.
Petryk N, Bultmann S, Bartke T, Defossez PA. Staying true to yourself: mechanisms of DNA methylation maintenance in mammals. Nucleic Acids Res. 2021 Apr 6;49(6):3020-3032. doi: 10.1093/nar/gkaa1154.
Baubec T, Defossez PA. Reading DNA Modifications. J Mol Biol. 2020 Feb 8:S0022-2836(20)30096-6. doi: 10.1016/j.jmb.2020.02.001.
Cornett EM, Ferry L, Defossez PA, Rothbart SB. Lysine Methylation Regulators Moonlighting outside the Epigenome. Mol Cell. 2019 Sep 19;75(6):1092-1101. doi: 10.1016/j.molcel.2019.08.026.
Naciri I, Laisné M, Ferry L, Bourmaud M, Gupta N, Di Carlo S, Huna A, Martin N, Peduto L, Bernard D, Kirsh O, Defossez PA. Genetic screens reveal mechanisms for the transcriptional regulation of tissue-specific genes in normal cells and tumors. Nucleic Acids Research, 07 February 2019 gkz080, https://doi.org/10.1093/nar/gkz080
Kori S, Ferry L, Matano S, Jimenji T, Kodera N, Tsusaka T, Matsumura R, Oda T, Sato M, Dohmae N, Ando T, Shinkai Y, Defossez PA, Arita K. Structure of the UHRF1 Tandem Tudor Domain Bound to a Methylated Non-histone Protein, LIG1, Reveals Rules for Binding and Regulation. Structure. 2018 Dec 12. pii: S0969-2126(18)30455-6. doi: 10.1016/j.str.2018.11.012.
Laisné M, Gupta N, Kirsh O, Pradahan S, Defossez PA. « Mechanisms of DNA Methyltransferase Recruitment in Mammals ». Genes 2018, 9(12), 617; doi: 10.3390/genes9120617
Marchal C, de Dieuleveult M, Saint-Ruf C, Guinot N, Ferry L, Olalla Saad ST, Lazarini M, Defossez PA, Miotto B. Depletion of ZBTB38 potentiates the effects of DNA demethylating agents in cancer cells via CDKN1C mRNA up-regulation. Oncogenesis. 2018 Oct 11;7(10):82. doi: 10.1038/s41389-018-0092-0.
Ma X, Warnier M, Raynard C, Ferrand M, Kirsh O, Defossez PA, Martin N, Bernard D. « The nuclear receptor RXRA controls cellular senescence by regulating calcium signaling ». Aging Cell. 2018 Sep 14:e12831. doi: 10.1111/acel.12831.
Miotto B, Marchal C, Adelmant G, Guinot N, Xie P, Marto JA, Zhang L, Defossez PA. « Stabilization of the methyl-CpG binding protein ZBTB38 by the deubiquitinase USP9X limits the occurrence and toxicity of oxidative stress in human cells. » Nucleic Acids Res. 2018 Feb 27. doi: 10.1093/nar/gky149.
Ferry L, Fournier A, Tsusaka T, Adelmant G, Shimazu T, Matano S, Kirsh O, Amouroux R, Dohmae N, Suzuki T, Filion GJ, Deng W, de Dieuleveult M, Fritsch L, Kudithipudi S, Jeltsch A, Leonhardt H, Hajkova P, Marto JA, Arita K, Shinkai Y, Defossez PA. “A histone mimic within DNA Ligase 1 links DNA replication and DNA remethylation: a revised model for the maintenance of DNA methylation by UHRF1”. Molecular Cell , 2017 Aug 17;67(4):550-565.e5. doi: 10.1016/j.molcel.2017.07.012. Epub 2017 Aug 10.
Naciri , A. Roussel-Gervais , P.-A. Defossez. O. Kirsh. “Nouvelles fonctions d’une protéine liant l’ADN méthylé dans le cancer”, Médecine/Sciences (nouvelle), Volume 33, Numéro 8-9, Août–Septembre 2017. https://doi.org/10.1051/medsci/20173308009
Roussel-Gervais , I. Naciri , O. Kirsh , L. Kasprzyk , G. Velasco , G. Grillo , P. Dubus, P.-A. Defossez..“Loss of the methyl-CpG binding protein ZBTB4 alters the mitotic checkpoint, increases aneuploidy, and promotes tumorigenesis”, Cancer Research, 2016 Nov 4. pii: canres.1181.2016
Santolini M, Sakakibara I, Gauthier M, Ribas-Aulinas F, Takahashi H, Sawasaki T, Mouly V, Concordet JP, Defossez PA, Hakim V, Maire P. « MyoD reprogramming requires Six1 and Six4 homeoproteins: genome-wide cis-regulatory module analysis. » Nucleic Acids Res. 2016 Oct 14;44(18):8621-8640. Epub 2016 Jun 14.
Ferrand, O. Kirsh, A. Griveau, D. Vindrieux, N. Martin, P.-A. Defossez, and D. Bernard, “Screening of a kinase library reveals novel pro-senescence kinases and their common NF-κB-dependent transcriptional program,” Aging (Albany NY), vol. 7, no. 11, pp. 986–1003, Nov. 2015.
Miotto, M. Chibi, P. Xie, S. Koundrioukoff, H. Moolman-Smook, D. Pugh, M. Debatisse, F. He, L. Zhang, and P.-A. Defossez, “The RBBP6/ZBTB38/MCM10 axis regulates DNA replication and common fragile site stability,” Cell Rep, vol. 7, no. 2, pp. 575–587, Apr. 2014.
Contact
Pierre-Antoine DEFOSSEZ
pierre-antoine.defossez@cnrs.fr
Membres

Pierre-Antoine Defossez
Team Leader | Deputy Director
pierre-antoine.defossez@univ-paris-diderot.fr
READ MOREFinancements




À lire aussi

Bienvenue à Léa
Léa rejoint l’équipe en tant qu’ingénieure d’études. Après un master en virologie, elle a travaillé à Strasbourg sur les virus de la vigne, puis sur la caractérisation de la dégradation des ARNm chez les plantes à l’Institut de biologie moléculaire des plantes (IBMP)....

Sophie Polo reçoit un financement Impulscience® de la Fondation Bettencourt Schueller
Sophie Polo a reçu un financement Impulscience® pour financer un projet de recherche sur l'établissement et le maintien du chromosome X inactif en réponse aux cassures de l'ADN. C'est une formidable nouvelle pour le laboratoire! Nous remercions la Fondation...
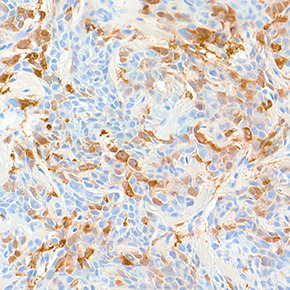
Quand retirer une étiquette de l’ADN stoppe la croissance tumorale
Chez les mammifères, la méthylation de l’ADN est une petite modification chimique qui agit comme une étiquette sur certaines bases de l’ADN, les cytosines. Elle n’altère pas la séquence mais participe au contrôle de l’activité des gènes et la stabilité du génome. Dans...

Bienvenue à Léa, nouvelle ingénieure d’études dans l’équipe!
Léa rejoint l'équipe en tant qu'ingénieure d'études. Titulaire d'un Master en biologie moléculaire et cellulaire de Sorbonne Université, Léa va contribuer à un projet de recherche visant à disséquer les mécanismes de maintien de la méthylation de l'ADN en réponse aux...