Transcription des séquences répétées centromériques
Un paradigme pour lier la transcription des séquences répétées de l’ADN à des effets moléculaires et cellulaires globaux
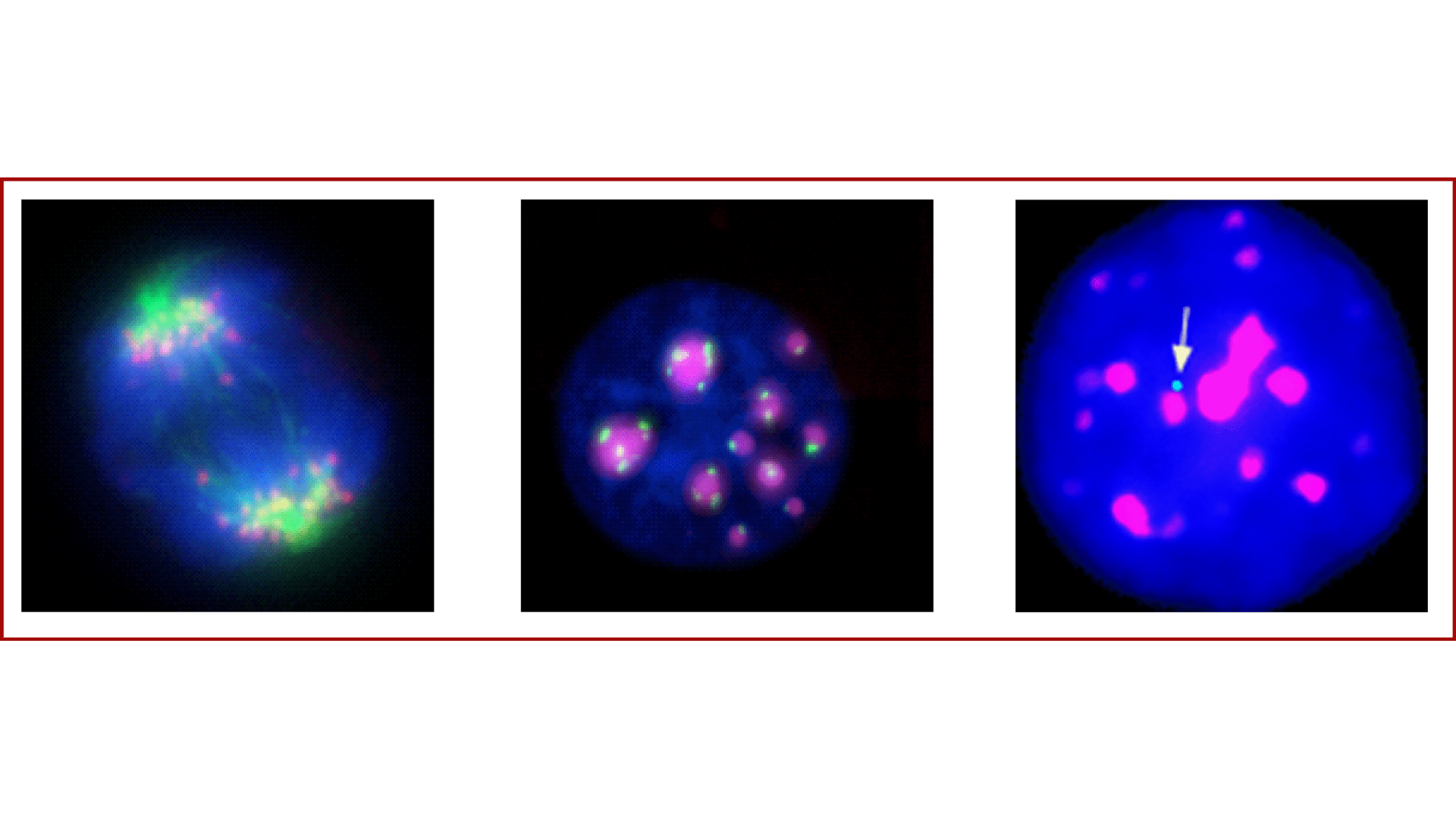
Cellules dans plusieurs phase du cycle cellulaire
© EDC
Les répétitions en tandem qui sous-tendent les régions centromériques ont un rôle structurel au niveau chromosomique, fournissant les plates-formes d’assemblage pour le kinétochore et l’arrimage du fuseau mitotique, mais aussi dans l’organisation fonctionnelle du noyau et le contrôle à longue distance de l’expression du génome.
Nous avons caractérisé les transcrits qui proviennent de répétitions centromériques murines et avons montré qu’elles sont essentielles pour l’identité et la fonction des centromères, alors que leur accumulation non contrôlée est causalement liée à une organisation nucléaire et à des phénotypes cellulaires perturbés. Cependant, nous avons montré que les conséquences dépendent largement des contextes cellulaires et génétiques. Dans les cellules primaires, la transcription accrue des répétitions centromériques fonctionne comme un détecteur de stress génotoxique et un déclencheur de mécanismes d’arrêt du cycle et de sauvegarde cellulaire. En revanche, dans des contextes où les points de contrôle qui dépendent de p53, cette accumulation conduit à une instabilité chromosomique, non sans rappeler les évènements oncogéniques.
Nous explorons le lien de causalité entre la transcription aberrante de séquences répétées et la perturbation des programmes moléculaires et cellulaires, ex vivo dans divers contextes cellulaires et génotypiques et in vivo dans des modèles murins. Nous interrogeons également les mécanismes qui conduisent à leur transcription dérégulée, avec un intérêt particulier pour la méthylation de l’ADN qui est étroitement liée au maintien de l’intégrité de ces séquences et, par conséquent, au maintien de la stabilité du génome. Finalement, nous cherchons à déchiffrer les fonctions cellulaires et les facteurs régulateurs qui sont perturbés par leur accumulation non programmée.
Publications sélectionnées:
- Hédouin S, Grillo G, Ivkovic I, Velasco G, Francastel C. CENP-A chromatin disassembly in stressed and senescent murine cells. Sci Rep. 2017 Feb 10;7:42520. doi: 10.1038/srep42520. PMID: 28186195
- Guillemin C, Francastel C. [Heterochromatin compartments and gene silencing: human hematopoietic differentiation as a model study]. Biol Aujourdhui. 2010;204(3):221-33. Review. French. PMID: 20950566
- Guillemin C, Maleszewska M, Guais A, Maës J, Rouyez MC, Yacia A, Fichelson S, Goodhardt M, Francastel C. Chromatin modifications in hematopoietic multipotent and committed progenitors are independent of gene subnuclear positioning relative to repressive compartments. Stem Cells. 2009 Jan;27(1):108-15. PMID: 18974210
- Ferri F, Bouzinba-Segard H, Velasco G, Hubé F, Francastel C. Non-coding murine centromeric transcripts associate with and potentiate Aurora B kinase. Nucleic Acids Res. 2009 Aug;37(15):5071-80. PMID: 19542185
À lire aussi

Bienvenue à Léa
Léa rejoint l’équipe en tant qu’ingénieure d’études. Après un master en virologie, elle a travaillé à Strasbourg sur les virus de la vigne, puis sur la caractérisation de la dégradation des ARNm chez les plantes à l’Institut de biologie moléculaire des plantes (IBMP)....

Sophie Polo reçoit un financement Impulscience® de la Fondation Bettencourt Schueller
Sophie Polo a reçu un financement Impulscience® pour financer un projet de recherche sur l'établissement et le maintien du chromosome X inactif en réponse aux cassures de l'ADN. C'est une formidable nouvelle pour le laboratoire! Nous remercions la Fondation...
Quand retirer une étiquette de l’ADN stoppe la croissance tumorale
Chez les mammifères, la méthylation de l’ADN est une petite modification chimique qui agit comme une étiquette sur certaines bases de l’ADN, les cytosines. Elle n’altère pas la séquence mais participe au contrôle de l’activité des gènes et la stabilité du génome. Dans...

Bienvenue à Léa, nouvelle ingénieure d’études dans l’équipe!
Léa rejoint l'équipe en tant qu'ingénieure d'études. Titulaire d'un Master en biologie moléculaire et cellulaire de Sorbonne Université, Léa va contribuer à un projet de recherche visant à disséquer les mécanismes de maintien de la méthylation de l'ADN en réponse aux...
